Resúmen
Los avances en el ámbito de la patología y cirugía realizados entre
1500 y 1750 sirvieron como base
para su desarrollo en los siglos XVIII y XIX, comprendiendo la
naturaleza y composición macroscópica y microscópica de los tumores
benignos y malignos. Desde entonces, los conceptos se volvieron
a enfocar desde el órgano al tejido y a la célula, afectando el
nacimiento de la histopatología que ha
dominado esta ciencia durante siglo y medio. Luego, cuando el segundo
milenio se acercaba a su fin,
nuevas y poderosas tecnologías comenzaron a forzar una nueva revisión
de las ideas alrededor de la
patología convencional, desde las enfermedades basadas en alteraciones
celulares hacia las enfermedades basadas en genes, pasando por el
estudio de moléculas individuales y su interacción. El auge de la
medicina de precisión comenzó en la década de 1980 con el desarrollo de
la inmunohistoquímica. Este
método permitió a los patólogos investigar rápidamente la expresión de
proteínas en piezas obtenidas
de muestras quirúrgicas. Estos niveles de expresión pronto serían
relevantes para las subclasificaciones
de los tumores que no eran accesibles por la microscopía óptica
clásica. Recientemente, la introducción de la patología molecular tuvo
un impacto positivo en el manejo de los pacientes con cáncer,
especialmente para seleccionar terapias dirigidas y permitir el uso de
técnicas como las biopsias líquidas
que establecieron un nuevo estándar para los regímenes de
monitorización continua durante el curso
de la enfermedad. Esta técnica permite la detección de las recurrencias
de la enfermedad antes que la
radiología, lo que permite adaptar las terapias con anticipación.
Además, también puede detectarse el
desarrollo de mutaciones somáticas asociadas con la resistencia a las
intervenciones realizadas.
Palabras clave: Patología; inmunohistoquímica; pruebas
moleculares; medicina de precisión.

¹ Sección Patología, Fundación para la Investigación Clínica y
Molecular Aplicada del Cáncer, Bogotá, Colombia.
² Departamento de
Patología, Universidad Nacional de Colombia, Bogotá, Colombia.
FROM IMMUNOHISTOCHEMISTRY OR THE “BROWN
REVOLUTION” TO THE DEVELOPMENT OF LIQUID
BIOPSY IN TUMOR PATHOLOGY
Abstract
Advances made between 1500 and 1750 in pathology and surgery served as
grounds for further progress in the 18th and 19th centuries in
understanding the nature and the macroscopic
and microscopic composition of benign and malignant tumors. Since then,
concepts were
re-focused from organ to tissue, to cell, ever smaller, affecting
histopathology’s birth that has
held sway in pathology for just a century and a half. Then, as the
second millennium drew to a
close, powerful new technologies began to force yet another revision of
conventional pathology ideas, from cell-based disease to gene-based
disease, to individual molecules and their
interplay. The rise of precision medicine began in the 1980s with the
development of immunohistochemistry. This method permitted pathologists
to quickly investigate various proteins’
expression on histological slides obtained from surgical specimens.
These expression levels
would soon become relevant for subclassifications of tumors that were
not accessible by light
microscopy alone. Recently, the introduction of molecular pathology
positively impacted cancer patient management, especially for selecting
targeted therapy and the use of techniques
like liquid biopsies that establish a new standard for continuous
monitoring regimens during
disease. This technique allows the detection of cancer recurrences
earlier than radiology, thus
allowing to adapt therapies earlier. Besides, the development of gene
mutations associated
with resistance to administered therapies might also be detected.
Keywords: Pathology; immunohistochemistry; molecular
tests; precision medicine.
Introducción
Hasta la primera mitad del siglo XX, los estudios
histopatológicos se basaban en la identificación de los
diferentes tipos celulares presentes en los tejidos, el
reconocimiento de patrones morfológicos característicos de los
diferentes procesos patológicos y en la detección de sustancias
diversas y de microorganismos,
mediante una amplia gama de técnicas de histoquímica (coloraciones
especiales), adaptadas de reacciones
químicas y de tinciones usadas en microbiología. A
partir del decenio de 1940 empezaron a introducirse
metodologías biotecnológicas. Inicialmente, en la medida que mejoraba
el conocimiento de la inmunología
empezaron a desarrollarse las pruebas de inmunofluorescencia e
inmunoenzimáticas, y una década después
con el mejor conocimiento de la biología de los ácidos nucleicos, se
desarrollaron la hibridación in situ, la
PCR y la secuenciación. Estas técnicas se han perfeccionado y
diversificado progresivamente, luego se han
automatizado gracias al desarrollo de instrumentos y
plataformas para garantizar su uso a gran escala de forma homogénea,
mejorándose con ello su difusión
con rigurosos estándares de calidad. La introducción
de estas técnicas en investigación biomédica y particularmente en
patología, ha contribuido a mejorar enormemente el conocimiento de
diferentes enfermedades,
entre las que se destacan las neoplasias. Su introducción progresiva al
diagnóstico patológico rutinario ha
permitido proporcionar mayor información adicional
sobre factores pronóstico y predictivos, contribuyéndose con ello a
mejorar notablemente la atención de
los pacientes.
Antecedentes
La inmunohistoquímica es una técnica utilizada en patología, basada en
el uso de anticuerpos como reactivos
altamente específicos para identificar una innumerable
gama de macromoléculas denominados marcadores
en cortes histológicos o preparaciones citológicas, por
medio de la visualización en el microscopio de reacciones
antígeno-anticuerpo. En su concepción más
amplia, incluye tanto las técnicas predecesoras de inmunofluorescencia
como las inmunoenzimáticas desarrolladas posteriormente.
Los antecedentes de estas pruebas se remontan al reconocimiento de las
propiedades de las inmunoglobulinas para identificar de manera
específica antígenos
diversos como parte de la respuesta inmune humoral,
para luego eliminarlos por medio de la activación del
complemento y de la quimiotaxis de fagocitos y luego
brindar inmunidad gracias a la activación clonal de los
linfocitos B de memoria y su diferenciación a plasmocitos, al volver a
ponerse el individuo en contacto con
dichos antígenos, produciendo nuevamente anticuerpos contra ellos a
gran escala.
Posteriormente fueron reconocidos los diferentes tipos
de inmunoglobulinas, sus funciones en la respuesta inmune – La IgM,
pentamérica que participa en la fase
inicial de la respuesta a agentes infecciosos, seguida
por la producción de IgG e IgA dimérica, ésta última
presente en superficies de las mucosas, la IgE en el reconocimiento de
alérgenos etc.- y en la generación de
las reacciones de hipersensibilidad, mediando el desarrollo de una
amplia gama de enfermedades. Luego
se develó su estructura, demostrándose en la cadena
pesada (fragmento Fc) las secuencias de aminoácidos
propias de cada tipo de inmunoglobulina y específicas de cada especie
animal en que se producen y en la
región hipervariable de las cadenas livianas, secuencias que les
confieren su capacidad para reconocer
específicamente innumerable variedad de antígenos y
sus epítopes. Luego, a partir de estos conocimientos
se definieron las bases de las pruebas serológicas para
determinar la exposición previa a diferentes agentes
infecciosos y se ideó la producción de sueros inoculando animales como
parte del tratamiento de diferentes
enfermedades. El origen de la inmunohistoquímica se
remonta a 1941, cuando Albert Coons (1912- 1978) en
la Universidad de Harvard ideó una prueba para reconocer de manera
específica neumococos en un microscopio de fluorescencia, mediante el
uso de anticuerpos
conjugados con fluoresceína (1).
Lógicamente el desarrollo de esta prueba requirió del
reconocimiento previo desde finales del siglo XIX de
las sustancias fluorescentes, moléculas que al ser expuestas a luz
ultravioleta tienen la capacidad de emitir
luz en el espectro visible: verde para la fluoresceína,
rojo para la rodamina etc., que hicieran merecedor del
Premio Nobel de Química en 1905 al químico alemán
Adolf von Baeyer (1835- 1917). Para principios del
siglo XX se habían descubierto una enorme gama de
estas sustancias y en 1913, los físicos Otto Heimstaedt
y Heinrich Lehmann, desarrollaron el microscopio de
fluorescencia, cuyo principio se basa en la utilización
de lámparas de mercurio, fuente de luz ultravioleta
para excitar los fluorocromos y un complejo sistema
de filtros para evitar la exposición del observador a la
luz ultravioleta y a la vez seleccionar la luz emitida por
cada fluorocromo para su visualización.
La técnica desarrollada inicialmente por Coons se
denomina inmunofluorescencia directa, considerando que cada anticuerpo
se encuentra acoplado en su
fragmento Fc a un fluorocromo. Posteriormente se
desarrolló la inmunofluorescencia indirecta, que permite obtener
preparaciones más limpias, económicas y
versátiles, en la que se utilizan dos anticuerpos obtenidos de dos
especies de animales diferentes, el primario
obtenido por ejemplo a partir de conejos inoculados
con el antígeno objeto de estudio, al cual reconocen
específicamente, mientras el secundario obtenido a
partir de la inoculación de animales de otra especie,
por ejemplo cabro con inmunoglobulinas de conejo y
que se conjugan con el agente fluorescente que reconoce la secuencia de
aminoácidos propia del fragmento
Fc de conejo (
Figura 1). Estas
técnicas siguen siendo
ampliamente utilizadas en diagnóstico patológico en
el estudio de enfermedades mediadas por mecanismos
inmunológicos, en cortes histológicos de tejido congelado,
particularmente de biopsias de piel y de riñón en
los cuales la epidermis, los vasos dérmicos y los glomérulos son
fácilmente identificables en el campo oscuro
al visualizarlos en el microscopio de fluorescencia.
Luego se intentó introducirlas para el estudio de otras
enfermedades como neoplasias pobremente diferenciadas para complementar
el estudio morfológico, precisando más específicamente su
diferenciación al identificar moléculas propias de su origen
histogenético. Sin
embargo, estos intentos inicialmente prometedores en
investigación, resultaron siendo infructuosos para su
uso a gran escala y en diagnóstico, por las dificultades
que planteaba en esa época disponer rutinariamente
de muestras de tejido congelado en instituciones de salud, por la
insuficiente disponibilidad de microscopios
de fluorescencia y sus dificultades para su uso, que llevarían
posteriormente a su perfeccionamiento con el
desarrollo de microscopios de epifluorescencia –con la
lámpara de mercurio dispuesta en la porción posterior
del equipo y un juego de prismas que dirigen la luz a
través del objetivo hacia la preparación histológica–,
más fáciles de utilizar. Por otra parte, la transitoriedad
de estas preparaciones, debido al agotamiento de los
fluorocromos (
efecto “fading”) para emitir luz en el
espectro visible luego de ser expuestos a la luz ultravioleta,
determinaba la necesidad de mantenerlos almacenados en la oscuridad, de
fotografiarse para obtener un registro duradero y por supuesto por la
imposibilidad
para obtener preparaciones adecuadas para interpretación en cortes de
tejidos fijados en formol e incluidos
en parafina, debido a que en el campo oscuro no es
posible discernir con precisión los rasgos morfológicos
para su adecuada interpretación (2, 3).
Más recientemente, gracias al desarrollo la hibridación
in-situ fluorescente y de la
microscopia confocal, ampliamente utilizada hoy en investigación
biomédica,
la inmunofluorescencia ha cobrado importancia muy
relevante, facilitando por ejemplo la identificación de
varios antígenos simultáneamente mediante anticuerpos marcados con
fluorocromos diferentes, permitiendo evaluar dinámicamente sus
interrelaciones por colocalización en cultivos celulares.
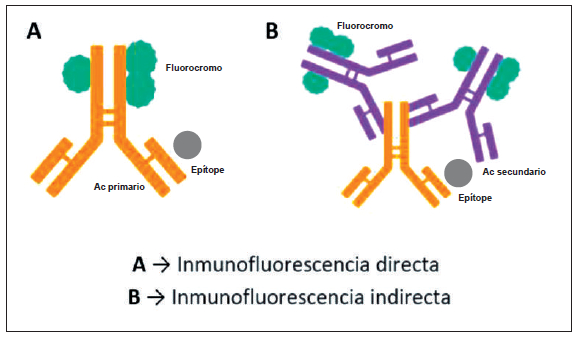
Figura 1. Representación esquemática de las técnicas de
inmunofluorescencia.
El advenimiento de las pruebas
inmunoenzimáticas
Debido al gran potencial que representaban los principios de la
inmunohistoquímica para caracterizar poblaciones celulares en
diferentes procesos patológicos
neoplásicos y no neoplásicos y para hacer más objetivo
y preciso el diagnóstico de tumores pobremente diferenciados, luego de
la imposibilidad de adaptar la inmunofluorescencia al estudio de
tejidos fijados en formol e incluidos en parafina, empezaron a
explorarse
otras alternativas para revelar la reacciones antígenoanticuerpo en los
tejidos. Al mismo tiempo, laboratorios y empresas de biotecnología
habían perfeccionado
las metodologías para obtener una gama creciente de
anticuerpos policlonales para identificar antígenos celulares, en la
medida que progresaba el conocimiento
de la biología celular y de la bioquímica. Los anticuerpos seguían
utilizándose en investigación usando las
pruebas de inmunofluorescencia.
Finalmente en 1967, Nakane y Pearce y otros grupos
de investigadores, encontraron como alternativa a los
fluorocromos acoplados a los anticuerpos, enzimas
que al interactuar con ciertos sustratos denominados
cromógenos generaban una reacción coloreada, que
permitía revelar las reacciones antígeno- anticuerpo en
cortes de tejidos (4-6). Estas enzimas se caracterizaban
por su estabilidad y permanecer activas a temperatura
ambiente en una solución tamponada a pH fisiológico
(7,1- 7,4); la primera enzima que garantizó resultados
reproducibles fue la peroxidasa obtenida de rábano
picante, utilizando como cromógeno la diaminobencidina que se colorea
de marrón, al cual el desarrollo
de las pruebas inmunoenzimáticas debe el nombre de
“revolución marrón” y con los que fue posible obtener
preparaciones permanentes contrastadas con tinción
nuclear de hematoxilina, inicialmente en cortes de tejidos frescos
congelados y en 1974 Taylor y Burns en la
Universidad de Oxford lograron su aplicación en cortes de tejidos
fijados en formol e incluidos en parafina
(7, 8), con rendimiento significativamente mejor que la
inmunofluorescencia (9). Otro cromógeno alternativo
debido al potencial carcinógeno de la manipulación de
diaminobenzidina pura, es el aminoetilcarbazol.
El camino para el desarrollo de las pruebas inmunoenzimáticas no estuvo
exento de dificultades, que motivaron
hacer modificaciones en el proceso histotecnológico y que
exigían la estandarización de cada anticuerpo con el uso
de cortes de tejidos con el antígeno a determinar usados
como controles positivos y controles negativos, correspondientes a
cortes del tejido estudiado a los que no se agregaba el anticuerpo
primario, para evaluar eventuales artificios y garantizar la adecuada
interpretación de las pruebas
inmunoenzimáticas (10-13).
La fijación tisular además de preservar la morfología
celular, debía entonces garantizar la preservación de
los determinantes antigénicos para su identificación
con los anticuerpos, lo cual llevó al abandono de gran
número de soluciones fijadoras, algunas usadas desde
el siglo XIX que brindaban una preservación morfológica excelente,
dentro de ellas se destacan el fijador
de Bouin preparado con ácidos acético y pícrico, los fijadores a base
de cloruro de mercurio, usado para
mejorar el detalle nuclear en las soluciones de Zencker, B5 y FMA
usados en nefro, hemato y dermatopatología. Incluso el formol al 10%,
fijador más ampliamente usado hasta entonces por su disponibilidad,
fácil preparación y bajo costo, debió modificarse, por
que el formaldehído al oxidarse espontáneamente se
transforma en ácido fórmico, acidificándose con ello
la solución fijadora, lo cual induce modificaciones
del punto isoeléctrico y cambios de la conformación
tridimensional de las proteínas que constituyen una
gran proporción de antígenos celulares, interfiriéndose así las
reacciones antígeno- anticuerpo. Para evitar
estos efectos se recomendó el uso universal de formol
tamponado neutro al 10%, utilizando tampones de
fosfato o tris, entre otros (14-16). Más recientemente
se ha propuesto la utilización de sustitutos del formol
tamponado, algunas comerciales para obtener mejores
resultados en estudios de inmunohistoquímica y moleculares (17-20).
Para preservar los anticuerpos concentrados deben
mantenerse congelados durante su transporte y almacenamiento, lo cual
requiere cadenas de frío.
Desde el inicio de su uso fue evidente la necesidad
de encontrar para cada anticuerpo la dilución más
apropiada en la que se produce la reacción antígeno anticuerpo. Para
obviar tener que diluir los anticuerpos concentrados, empezaron a
comercializarse
anticuerpos prediluidos que pueden almacenarse refrigerados a 4 grados
centígrados, pero su tiempo de
uso es limitado, requiriéndose entonces, prolongar el
tiempo de incubación para seguir obteniendo resultados adecuados.
Inicialmente empezaron a usarse las técnicas directa e
indirecta, similares a las utilizadas en inmunofluorescencia, pero
pronto fue evidente la necesidad de desarrollar complejos
inmunoenzimáticos para minimizar
artificios como la inespecificidad de fondo por la precipitación del
cromógeno, así como para amplificar la
reacción antígeno anticuerpo y mejorar la sensibilidad
de las pruebas. Los primeros complejos que se desarrollaron fueron la
peroxidasa antiperoxidasa (9, 21, 22),
la avidina biotina peroxidasa y posteriormente la estreptavidina
biotina peroxidasa (23, 24) (
Figura 2),
con
las cuales empezó la difusión global de la técnica para
diagnóstico a finales del decenio de los años ochenta.
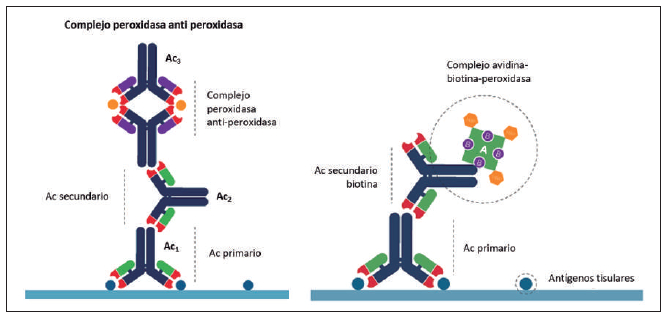
Figura 2. Representación esquemática de las técnicas de
inmunohistoquímica: Complejos inmunoenzimáticos
La actividad de peroxidasas endógenas en algunos tipos celulares como
los hematíes y fagocitos fue otro
factor determinante de artificios por dar lugar a reacciones
inespecíficas con los cromógenos, que determinaron muy rápidamente el
desarrollo de alternativas
para su bloqueo, lo cual se logra con el pretratamiento
de los cortes de tejido con soluciones de peróxido de
hidrógeno (25-27). Otra fuente de inespecificidad de
fondo identificada inicialmente, estaba determinada
por la presencia de biotina en los cortes de tejido hepático en el cual
se recomendaba usar otro complejo.
Como alternativa a la peroxidasa se introdujeron otras
enzimas como la fosfatasa alcalina (28, 29) y la glucosa oxidasa (30)
que interactuaban con los cromógenos
fast red y fast blue para la primera y tetrazolium blue
para la segunda, de tonalidades diferentes que ofrecían
alternativas por ejemplo para el estudio de proliferaciones
melanocíticas con pigmento melánico, difíciles
de discernir usando peroxidasa y diamino bencidina
y además se convertían en una opción para la identificación simultánea
de dos marcadores identificables
con cromógenos diferentes (31). Para ello también se
desarrollaron técnicas de inmunomarcación usando
oro y plata conocidas como immunogold (32, 33), que
evitan confusiones con la reactividad de fondo por peroxidasa endógena
y se usan en inmunoelectromicroscopía como una adaptación de la
inmunohistoquímica
para la identificación ultraestructural de marcadores,
utilizando oro coloidal acoplado a los anticuerpos en
investigación (34, 35).
El efecto de la fijación del formol reconocido desde
mediados del siglo XX es debido a la producción de
puentes de hidrógeno con las diferentes macromoléculas y entre ellas,
el cual se acentúa con el tiempo de fijación (16, 36, 37), lo cual
puede interferir la detección
de los antígenos por un efecto de enmascaramiento, el
cual empezó a contrarrestarse con el pretratamiento de
los cortes de tejido con enzimas como tripsina, pronasa y quimotripsina
(38, 39).
Otra dificultad que debió enfrentarse fue el desprendimiento de los
tejidos de las láminas portaobjeto al aplicar estas pruebas, debido a
la duración de su exposición
al medio acuoso en que se desarrollan la incubación
con los anticuerpos, complejos inmunoenzimáticos, el
bloqueo de la peroxidasa endógena y los lavados con
soluciones tamponadas entre los diferentes pasos antes
de aplicar la coloración nuclear de contraste, para lo
cual empezaron a utilizarse láminas pretratadas con
soluciones de y gelatina y poli L lisina y luego empezaron a
comercializarse láminas cargadas.
La especificidad de los anticuerpos utilizados en estas
técnicas mejoró notoriamente con la introducción de la
técnica del hibridoma que hiciera merecedores del premio
Nobel en 1984 a Kohler, Milstein y Jerne, para la producción in vitro
de anticuerpos, que se basa en la fusión en
cultivos celulares de células neoplásicas de plasmocitomas con células
B estimuladas con un antígeno para la
producción de anticuerpos monoclonales. Estos a diferencia de los
anticuerpos policlonales obtenidos inoculando
animales con antígenos que reconocen varios determinantes antigénicos,
identifican un epítope específico (40).
Todas estas modificaciones se desarrollaron realizando múltiples
ensayos a lo largo de casi dos decenios
hasta finales de los años 70, permitiendo algo más de
una década después, la introducción de las técnicas
inmunoenzimáticas para diagnóstico en un número
creciente de laboratorios de patología, inicialmente en
países del primer mundo y luego en la medida en que
más patólogos y laboratoristas se familiarizaban con su
uso, su expansión permitió la reducción de sus costos
y su uso se difundió globalmente. Con la aparición de
estos métodos se generó una nueva área de la industria
biotecnológica, que incluso dio lugar a la aparición de
conglomerados industriales, donde empezaron a producirse masivamente
los insumos requeridos para su
creciente aplicación en los laboratorios de patología,
que debieron dedicar áreas de sus instalaciones cada
vez mayores. Estos desarrollos determinaron el requerimiento a la
industria de estrictos controles de calidad
para garantizar la especificidad de los anticuerpos y el
adecuado funcionamiento de los “kits” de detección,
brindar la información pertinente a los usuarios sobre
las características de los reactivos e instrucciones detalladas para
almacenamiento, su uso (40) y también la
adopción de controles de calidad en cada laboratorio
llevando registros correspondientes sobre las propiedades de los
reactivos y la aplicación de las pruebas
para garantizar los mejores resultados (10, 11, 13, 41-
44), desde la fase preanalítica con recomendaciones
para el mejor manejo inicial de los diferentes tipos de
muestras, para garantizar la aplicación de pruebas de
inmunohistoquímica y luego moleculares (45), hasta
la fase posanalítica con indicaciones para garantizar
que el archivo del material histológico se realice en las
mejores condiciones por tiempos prolongados, debido
a la observación de degradación de determinantes antigénicas y otras
macromoléculas en cortes de tejidos
incluidos en parafina (46, 47), para beneficio de los
pacientes por el desarrollo de nuevos tratamientos basados en la
aplicación de pruebas asociadas.
Desarrollos posteriores
Se desarrollaron nuevos métodos de recuperación antigénica para
optimizar el uso de las preparaciones a partir de tejidos fijados en
formol tamponado e incluidos
en parafina, con el fin de contrarrestar la formación de
puentes de hidrógeno con el formaldehido y entre las
macromoléculas usando soluciones tamponadas de citrato en medio ácido y
de EDTA en medio alcalino de
acuerdo con las propiedades de los antígenos y de los
anticuerpos y sometiéndolas a calor, utilizando hornos
microondas o recipientes con presión provistos de termostatos para
mantener temperaturas estables (48-52).
Para mejorar la sensibilidad de las pruebas, se diseñaron alternativas
a los complejos inmunoenzimáticos,
desarrollando polímeros de dextrano o multímeros
acoplados a los anticuerpos secundarios (
Figura 3),
con los cuales la proporción de moléculas de enzimassustrato por
anticuerpo aumentó notoriamente (50,
53-58).
Un número creciente de marcadores, además de su
identificación cualitativa requiere de la cuantificación
de su expresión, para lo cual se idearon escalas análogo- visuales que
permiten hacer al patólogo una valoración semicuantitativa de rangos de
expresión que
empezaron a utilizarse rutinariamente, por ejemplo
con el sistema Allred para la evaluación de los receptores hormonales
(58- 59), para el valorar la extensión
de la expresión de Her 2 (59, 60) y Ki- 67 en cáncer de mama, o en los
tumores neuroendocrinos bien diferenciados. Con el fin de hacer una
valoración más
precisa, particularmente en casos con expresión cercana a puntos de
corte para definir pronóstico o tomar
decisiones terapéuticas se ha introducido la utilización
de analizadores de imagen (61, 62), herramientas de
software utilizadas inicialmente en imágenes obtenidas con cámaras de
video acopladas a los microscopios ópticos y más recientemente
incorporadas a los
nuevos microscopios digitales.
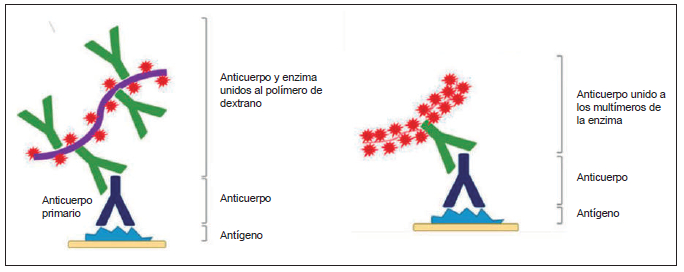
Figura 3. Representación esquemática de los complejos
inmunoenzimáticos con polímeros y multímeros.
Inicialmente las pruebas de inmunohistoquímica se
realizaban manualmente, posteriormente en la medida
en que aumentaba el volumen de este tipo de pruebas
en los laboratorios de patología, empezaron a desarrollarse equipos
para su automatización, inicialmente
similares a los equipos diseñados para histoquímica y
luego con especificaciones que permitían homogenizar su realización o
para mejorar la reproducibilidad,
garantizando que no se omitieran pasos durante la realización de la
prueba o que los diferentes anticuerpos,
reactivos y tampones con los que se tratan los cortes
de tejido fueran completamente cubiertos por estos
durante todo el desarrollo de la prueba, con lo cual
además de mejorarse los estándares de calidad (63-65),
se optimizo el uso del tiempo del personal de los laboratorios,
lógicamente estas adaptaciones aumentaron los costos en laboratorios de
bajo volumen y las
industrias de biotecnología empezaron a utilizar los
modelos de negocio que se habían implementado en
los laboratorios clínicos, asumiendo la dotación de los
laboratorios con equipos en comodato a cambio de la
fidelización de los laboratorios con los anticuerpos y
demás kits de reactivos de su marca.
Con el advenimiento de la inmunoterapia, ha surgido
más recientemente la necesidad de conocer más a fondo la respuesta
inmune a los tumores, la relación de las
células tumorales con los linfocitos intratumorales y el
estroma tumoral con el fin de diseñar alternativas de tratamiento (66,
67). En este contexto se ha propuesto definir la composición de los
infiltrados linfoides asociados
a los tumores haciendo un “
inmunoscore”,
identificando
marcadores asociados a respuestas inmunes eficientes o
a su supresión. Cada marcador se identifica por separado, o a lo sumo
por pares, utilizando dos enzimas con
sus respectivos cromógenos para revelarlos, pero con el
fin de evaluar simultáneamente varios marcadores, tomando como
referencia la experiencia de la citometría
de flujo en el estudio de las leucemias, han empezado
a desarrollarse sistemas de inmunohistoquímica multidetección (68). Su
análisis, sin embargo, requiere además del uso de analizadores de
imagen para determinar
con precisión las proporciones de los marcadores, de
programas de inteligencia artificial para evaluar las relaciones entre
las señales de las diferentes células caracterizadas con esta
metodología (69). Estas herramientas
por ahora tienen un prometedor campo de aplicación
en investigación, pero pronto se prevé, empezarán a migrar al
diagnóstico.
Evolución de la Utilización de las Pruebas
Inmunoenzimáticas en Diagnóstico
El desarrollo de las pruebas inmunoenzimáticas generó inicialmente
enormes expectativas en el ejercicio de
la patología, considerando que su uso permitiría impactar el
diagnóstico en situaciones en las que la subjetividad del análisis de
imágenes generaba grandes dificultades para efectuar diagnóstico
específico, incluso
llegó a pensarse que la inmunohistoquímica podría
reemplazar el análisis de patrones morfológicos en
patología tumoral por la identificación de marcadores
propios de cada tipo celular. De hecho, inicialmente se
creía que los diferentes antígenos para los cuales se desarrollaban
anticuerpos eran específicos de cada tipo
celular, sin embargo, su uso creciente demostró muy
pronto la ubicuidad de un número significativo de estos marcadores en
variable número de tipos celulares,
generándose con ello decepción, incluso el surgimiento de detractores
de la prometedora técnica, lo cual
obligó a replantear su utilización, siempre tomando como punto de
partida la identificación de los rasgos
morfológicos del análisis patológico clásico, para la selección de
paneles de marcadores para establecer diagnósticos diferenciales,
identificando la co-expresión de
algunos marcadores y la ausencia de otros antígenos,
definiéndose con ello el uso de perfiles de expresión
de marcadores que permitieron establecer con mayor
precisión diagnósticos diferenciales.
Un aspecto relevante del uso de la inmunohistoquímica
en el estudio de procesos patológicos, particularmente
neoplásicos ha sido la confirmación de los criterios de
clasificación histopatológica con base en rasgos morfológicos, sólo
ocasionalmente su uso ha determinado
un replanteamiento radical de las clasificaciones morfológicas. Por el
contrario, la identificación de perfiles
de expresión de marcadores ha permitido separar mas
claramente entidades nosológicas o identificar subcategorías
adicionales de procesos patológicos con comportamiento biológico
diferente. El ejemplo mas llamativo
de este impacto ha sido la hematopatología, área pionera en el uso de
la IHQ, que determinó muy pronto
grandes aportes al sistema de clasificación vigente de
Rappaport para los
linfomas no Hodgkin (70): su uso
determinó el planteamiento de la clasificación de Lukes
y Collins en Norteamérica, basado en criterios de inmunofenotipo
lukes(71). La aparición simultánea de
nuevas nomenclaturas y el uso por diferentes grupos de
investigadores en el mundo de anticuerpos que identificaban los mismos
marcadores pero denominados con
nombres diferentes, generaron una gran confusión, que
se solucionó por una parte con la creación de la nomenclatura de
consenso el “Working Formulation” para homologar los sistemas de
clasificación (72) y la creación
de una nomenclatura internacional para denominar los
marcadores usados en hematopatología el “
cluster differentiation o CD” (73,
74).
La creciente aparición de nuevos marcadores, determinó un notorio
incremento de la literatura para recomendar su aplicación en
situaciones específicas y
la formulación de algoritmos para la aplicación de los
diferentes paneles de anticuerpos para facilitar su utilización
racional en el diagnóstico de las neoplasias.
Por ejemplo, para el diagnóstico de tumores indiferenciados de origen
desconocido, de acuerdo con sus
características morfológicas, se plantea el uso de un
panel inicial para determinar la estirpe celular, usando
marcadores de amplia distribución en células epiteliales,
hematolinfoides, mesenquimales y por supuesto
los melanomas que por su morfología variable simulan neoplasias de
otros orígenes. Una vez se establece
la línea de diferenciación se solicita un panel adicional
para determinar con mayor precisión el origen del tumor con base en la
identificación de perfiles de marcación característicos. Para el caso
de tumores epiteliales
metastásicos de origen desconocido los algoritmos recomiendan el uso
inicial de paneles de origen mas probable de acuerdo con el órgano
afectado: ganglios linfáticos de diferentes localizaciones, hígado,
pulmón,
hueso, sistema nervioso, etc. (75, 76).
En algunos órganos como pulmón e hígado, donde son
más frecuentes los tumores metastásicos y estos comparten rasgos
morfológicos con algunos tumores primarios particularmente del tipo
adenocarcinoma, los
cuales tienen un tratamiento diferente, la inmunohistoquímica permite
la exclusión de metástasis mediante
paneles con marcadores que permiten definir con precisión neoplasias
primarias. En pulmón, este aspecto
se hizo especialmente relevante con el surgimiento de
tratamientos con moléculas pequeñas y anticuerpos
monoclonales efectivos, dirigidos a blancos y más recientemente con el
advenimiento de la inmunoterapia,
que empezaron a mejorar significativamente el tiempo sobrevida global y
libre de recaída de los pacientes,
lo cual determinó el reto de optimizar las limitadas
muestras de biopsias transbronquiales y por punción,
para que además de precisar el diagnóstico sirvieran
posteriormente para efectuar los estudios moleculares
pertinentes para definir los blancos terapéuticos (77).
Aplicaciones en Medicina
Personalizada
El otro aspecto en el cual la inmunohistoquímica empezó a tener un
papel muy relevante fue el teranóstico
propiamente dicho (78-80), término proveniente de la
industria farmacéutica, acuñado por John Funkhouser
“
CEO” de Pharma Netics, en
2007 (83, 84), aunque
su práctica precede su origen lingüístico, considerando
que su uso a gran escala se inició en el decenio de los
80 para el tratamiento hormonal del cáncer de seno
con tamoxifen dirigido a los receptores de estrógenos,
inicialmente determinados por métodos bioquímicos
que fueron exitosamente reemplazados por las novedosas técnicas de
inmunohistoquímica (81-83).
En el decenio de 1990, hubo otro descubrimiento sorprendente que
permitiría esclarecer el misterioso origen
de los tumores estromales gastrointestinales, más frecuentes en
estómago, los cuales hasta entonces solían
diagnosticarse como leiomiomas, leiomiosarcomas o
tumores derivados de la vaina neural, por su variada
morfología fusocelular o epitelioide con rasgos que sugerían estos
orígenes, no obstante ser negativos para
marcadores de músculo liso y nervio periférico, hasta
que se identificó la expresión de C-Kit o CD117 en las
células tumorales, demostrándose al mismo tiempo su
origen a partir de las células intersticiales de Cajal que
regulan el peristaltismo en el tubo digestivo y determinando al mismo
tiempo con ello un blanco terapéutico
para imatinib (glivec) (88, 89), con el cual se habían
obtenido excelentes resultados en el tratamiento de la
leucemia mieloide crónica, convirtiéndola de una neoplasia indolente
que tarde o temprano cobraba la vida
de los pacientes afectados por ella a convertirse en una
enfermedad crónica con una prolongada vida libre de
enfermedad (84, 85).
Por esa misma época, fue evidente la presencia en un
grupo de carcinomas mamarios con pobre pronóstico,
caracterizados por la significativa amplificación del
factor de crecimiento epidérmico de tipo II o Her 2
neu, que da lugar a una proliferación celular incontrolada por la
activación de una vía tirosina quinasa (86),
para el cual se desarrolló una prueba de inmunohistoquímica para
determinar su sobreexpresión, otra de
hibridación in-situ para demostrar la amplificación del
gene en los casos ambiguos por inmunohistoquímica
(59, 60, 87) y un anticuerpo monoclonal humanizado,
el trastuzumab (Herceptin) para tratamiento dirigido
contra este blanco, con el cual empezaron a obtenerse
excelentes resultados (88).
Para los linfomas no Hodgkin B, empezó a desarrollarse una alternativa
similar con el rituximab (Rituxan),
un anticuerpo monoclonal humanizado dirigido contra CD20, el cual
empezó a incorporarse a los esquemas de quimioterapia o conjugándolo
con radioisótopos (89).
Con el progresivo desarrollo de la medicina personalizada en oncología,
recientemente el número de
blancos terapéuticos susceptibles de identificarse por
inmunohistoquímica en diferentes tipos de neoplasias
se ha incrementado notoriamente, convirtiéndose en
una actividad rutinaria creciente en los laboratorios
de patología en todo el mundo. Para ello se han desarrollado los “
Companion Test”, pruebas de
inmunohistoquímica diseñadas por la industria farmacéutica y
aprobados por las agencias estatales reguladoras para
seleccionar específicamente los pacientes candidatos a
terapia personalizada dirigida a blancos terapéuticos
e inmunoterapia, utilizando clones de anticuerpos dirigidos a epítopes
específicos, revelados con “kits” de
detección definidos y procesados en plataformas definidas, para
garantizar resultados homogéneos con altos estándares de calidad que
incluyen la capacitación
de patólogos para la interpretación de las pruebas en
laboratorios de referencia (79, 80, 90). Desde la descripción de las
primeras técnicas inmunoenzimáticas
su aplicación en investigación y diagnóstico ha aumentado enormemente
como lo muestra la litertura (91).
Hibridación in-situ
El primer campo de aplicación rutinaria de la citogenética en la
patología fueron las leucemias, inicialmente con
la identificación del cromosoma Filadelfia en leucemia
mieloide crónica (92) y luego con la descripción creciente de otras
anormalidades cromosómicas (duplicaciones, translocaciones, genes de
fusión, deleciones), cuya
identificación se incorporó progresivamente con fines de
diagnóstico y pronóstico en hematopatología (93) y más
recientemente en el estudio de tumores sólidos.
El conocimiento progresivo sobre la estructura de los
ácidos nucleicos y la biología de los procesos de replicación,
transcripción y síntesis proteica a partir de los
trabajos de Watson y Creek (94, 95), que les hiciera
merecedores del premio nobel de Medicina y Fisiología en 1962, dio
lugar a la aparición de nuevas metodologías para su aplicación inicial
en investigación
biomédica y luego en diagnóstico. El desarrollo de las
técnicas de citogenética y de hibridación
in situ se aceleró notoriamente
desde los años 70, dando lugar al desarrollo de una amplia gama de
pruebas diagnósticas
con una creciente aplicación en genética y patología
molecular durante el siglo XXI (96-100), determinando que, desde su
descripción, aumentara de manera
exponencial su aplicación en investigación, como lo
muestra la literatura (24).
En 1969 Gall y Pardue (101) y John, Birnstiel y colaboradores (102),
desarrollaron simultáneamente la
prueba de hibridación in- situ en preparaciones citológicas, cuyo
principio se basa en la identificación de
secuencias de nucleótidos con una sonda complementaria marcada
inicialmente con isótopos radioactivos
mediante autorradiografía, luego en preparaciones
histológicas (103) y su aplicación empezó una década
después (104), cuando se desarrollaron las pruebas de
hibridación in situ fluorescente (FISH por su acrónimo
en inglés), directas con el fluorocromo conjugado a la
sonda e indirectas cuando se conjugan con un hapteno o etiqueta como
digoxigenina o biotina, que en un
paso posterior son reconocidos respectivamente por
un anticuerpo antidigoxigenina o avidina acoplados
con el fluorocromo o una sustancia quimioluminicente. Las sondas por su
parte pueden ser de DNA monocatenario o RNA (96-100) (
Figura 4).
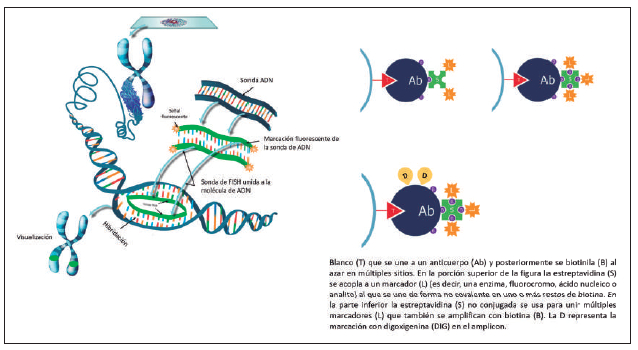
Figura 4. Representación esquemática de la hibridación in-situ.
Con estas pruebas pueden identificarse secuencias de
DNA y RNA, estas últimas requieren de un óptimo manejo de los tejidos
en la fase preanalítica para garantizar
su preservación debido a su labilidad; al igual para que
las pruebas inmunoenzimáticas, empezaron a realizarse
en láminas pretaratadas con gelatina o poli L lisina y
luego en láminas cargadas para evitar el desprendimiento de los cortes
por requerir múltiples pasos en medio
acuoso, entre los que se destacan un pretratamiento con
proteasas, generalmente proteinasa K, la denaturación
del DNA para separar las dos cadenas, seguida por la
hibridación que generalmente se hace durante la noche
y lavados con soluciones tamponadas entre los diferentes pasos; para
facilitar la identificación de los núcleos
se utilizan fluorocromos con gran afinidad por el DNA
bicatenario como el DAPI (4’,6-diamino -2-fenilindol),
que brindan una coloración azul de contraste (105,
106). Para la interpretación se analiza un número representativo de
células o núcleos identificando las señales
fluorescentes marcadas.
El FISH, ampliamente extendido hoy en investigación,
diagnóstico y en la evaluación de factores de pronóstico y predictivos,
adolece de los mismos inconvenientes
de la inmunofluorescencia: preparaciones transitorias
por el efecto de agotamiento (“
fading”)
de los fluorocromos que requieren fotografías para su documentación y
archivo y la necesidad de utilizar microscopio
de fluorescencia. Para obviar estas dificultades posteriormente se
desarrolló la hibridación in- situ cromogénica (CISH), con base en
algunas adaptaciones
procedentes del desarrollo de la inmunohistoquímica,
como el uso de complejos inmunoenzimáticos o plata, con las cuales
además de obtenerse preparaciones
permanentes. con preservación de los rasgos morfológicos resaltados con
la coloración de hematoxilina
usada para dar contraste nuclear, se analizan en microscopios
convencionales (107, 108).
Para facilitar la evaluación se desarrollaron también
pruebas duales, con las cuales además de las secuencias problema a
identificar, suelen evaluarse simultáneamente sondas marcadas con otro
fluorocromo o
cromógeno de secuencias de los centrómeros de los
cromosomas, donde se encuentran los genes que se
están evaluando, con la finalidad de facilitar el análisis (109).
Adicionalmente se han desarrollado pruebas
multicolor, más utilizadas en estudios citogenéticos.
La hibridación
in situ es una
prueba accesible a los
laboratorios de patología, Inicialmente empezó a
utilizarse en preparaciones citológicas, mielogramas,
extendidos de sangre periférica, improntas, cortes de
tejidos frescos congelados, luego en tejidos fijados en
formol e incluidos en parafina; también es usado en
investigación en cultivos celulares. Desde su descripción, esta
metodología se ha difundido masivamente
en el estudio de múltiples procesos patológicos dentro
de los que se destacan neoplasias, enfermedades infecciosas etc. En
hematología, empezó a utilizarse para
el diagnóstico rutinario de leucemias, luego de linfomas para la
identificación de agentes virales como el
virus de Epstein Barr, que por integrarse al genoma
frecuentemente no puede detectarse con la inmunohistoquímica para LMP1,
mientras la hibridación in situ
para EBER es muy sensible (110); es una herramienta
muy útil en la identificación de rearreglos en linfomas
no Hodgkin para definir diagnósticos diferenciales y
factores de pronóstico (111-114). Su aplicación ha contribuido al
desarrollo de la medicina personalizada en
el estudio de tumores sólidos para la identificación de
blancos terapéuticos: entre las primeras aplicaciones
del FISH y el CISH, se encuentran la amplificación de
Her2-neu en casos de cáncer de mama con resultados
ambiguos o equívocos en la evaluación de la expresión
del marcador por inmunohistoquímica (87) de translocaciones de EML4-ALK
en adenocarcinoma de pulmón (115, 116), detección de virus de papiloma
humano y virus de Epstein Barr en carcinomas de cabeza y
cuello y rearreglos en neoplasias de glándulas salivares
(117), rearreglos para el diagnóstico diferencial de melanomas (118) y
sarcomas (119) etc.
Para facilitar el proceso se han diseñado equipos, plataformas para su
automatización y analizadores de
imagen para su interpretación, mejorándose con ello
la reproducibilidad y otros estándares de calidad (120).
Microdisección
En 1975 Frederik Sanger (1918- 2013) desarrolló en la
Universidad de Cambridge la metodología de secuenciación de ácidos
nucleicos (121), que le hiciera merecedor en 1980 a un segundo premio
Nobel de Química,
la cual pese a haberse descrito variantes y automatizado dando lugar a
plataformas muy robustas, continua
siendo el estándar de oro de estas técnicas, gracias a las
cuales ha mejorado enormemente el conocimiento del
genoma, contribuyendo en forma decisiva la investigación biomédica y
más recientemente al diagnóstico de
un sinnúmero de enfermedades (122).
En 1988 Kari Mullis (1944- 2019) describió la reacción
en cadena de la polimerasa utilizando la DNA polimerasa aislada de
Thermus aquaticus, la cual mantiene
su
actividad a temperaturas superiores a 75o
C (123-125),
que también le hiciera merecedor del premio nobel de
química en 1993, su posterior automatización y la descripción de
variantes (anidada, reversa, en tiempo real,
multiplex, digital) permitiría contar con una herramienta muy eficiente
y actualmente indispensable para el estudio del genoma en investigación
biomédica.
Inicialmente estas metodologías demostraron su utilidad en patología
tumoral para el estudio en muestras
de tejido fresco congelado y posteriormente empezaron a aplicarse a
muestras de tejidos fijados en formol
e incluidos en parafina, con resultados progresivamente mejores en
estudios genómicos y de expresión génica de tumores sólidos. En la
actualidad, la principal
fuente para estos estudios de diagnóstico y teranóstico
son los tejidos incluidos en parafina disponibles en los
archivos de los laboratorios de patología y los estudios
histopatológicos continúan siendo la base para la selección de material
histológico para la adecuada aplicación de pruebas moleculares (126).
Inicialmente se
presentaron algunas dificultades para su análisis en los
resultados, relacionadas con la variable representación
de los tumores en las muestras por su agotamiento en
los bloques de parafina, por la presencia de necrosis
extensa o de áreas con preservación deficiente, que se
obviaron con el desarrollo de la microdisección de tejidos (127-130),
técnica que se basa en la selección de
los bloques con mejor representación de los tumores
con base en la observación de las preparaciones histológicas
correspondientes, de los cuales se obtienen
nuevos cortes más gruesos que se colorean con eosina
y de los cuales se extrae manualmente el tejido con
adecuada representación de los tumores, para colocarlos en tubos
eppendorf y cuando las muestras son muy
limitadas se extrae el tejido disponible del bloque. A
partir de este material, luego de la desparafinización,
se realiza la extracción de los ácidos nucleicos y posteriormente su
amplificación mediante PCR y su secuenciación (131, 132). Para algunos
estudios de investigación, sin embargo, las técnicas manuales no
garantizan
la obtención de muestras sin elementos tisulares contaminantes, por lo
cual posteriormente se desarrollo la
técnica de microdisección láser (133, 134), que se basa
en la observación en un microscopio provisto de una
cámara de video de la lámina histológica para luego
delimitar el área de tejido seleccionado para estudio,
el cual se corta mediante láser y luego se transfiere a
un tubo eppendorf. L a precisión de ésta metodología
incluso permite obtener células aisladas (135) para su
estudio molecular, con las cuales ha sido posible develar la patogenia
de neoplasias como el linfoma de Hodgkin caracterizado por la presencia
de células de Reed
Sterneberg o sus variantes dispersas en una población
predominante de células reactivas (136, 137).
Las técnicas de microdisección, han sido incorporadas
para la evaluación sistemática de factores predictivos
en un número creciente de tumores sólidos avanzados,
para terapia personalizada dirigida a blancos específicos con
anticuerpos monoclonales y moléculas pequeñas, con una significativa
mejoría de los resultados en
sobrevida global y libre de recaída en tumores en los
que antes del advenimiento de estos tratamientos, los
resultados eran muy limitados.
Biopsia Líquida
La identificación de células tumorales circulantes se
convirtió en un objetivo importante del estudio de la
biología de las metástasis, luego ocurriría lo mismo con
fragmentos circulantes de las células neoplásicas denominados exosomas,
resultantes de procesos de apoptosis, para los cuales se postuló un
papel importante en
el preacondicionamiento de los nichos metastásicos.
Luego hubo un enfoque específico sobre fracciones de
ácidos nucleicos libres como DNA y diferentes tipos de
RNAs de células tumorales en la circulación (
Figura 5);
en su sentido más amplio, el concepto de biopsia líquida, término
acuñado en 2010 (138), aunque, el concepto empezara a usarse algunos
años antes (139), abarca el
estudio además de la sangre de otros líquidos corporales
como pleural, peritoneal, cefalorraquídeo, orina etc. El
estudio de estos elementos aporta información adicional sobre la
heterogeneidad tumoral, por presentar frecuentemente características
moleculares diferentes de
los tumores primarios e incluso de las metástasis (138-
140). El limitado número de estos elementos circulantes
planteó un importante reto para su estudio, que llevó
inicialmente al desarrollo de metodologías para su aislamiento
diferencial de elementos circulantes similares
provenientes de elementos celulares no neoplásicos, que
se basa en el uso de métodos inmunocitoquímicos para
aislar los mediante anticuerpos dirigidos contra antígenos de células
epiteliales (141, 142).
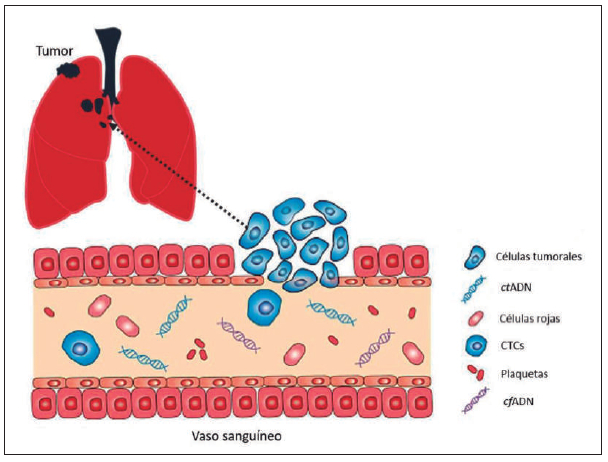
Figura 5. Representación esquemática de los elementos que se
analizan en
una biopsia líquida provenientes de una neoplasia pulmonar.
La evaluación de pacientes con recidivas recurrentes de
neoplasias que habitualmente requieren de la obtención
de varias biopsias para su demostración y la necesidad
de estudiar factores asociados a resistencia al tratamiento,
encontraría en el estudio de estos elementos circulantes una
alternativa, que dio lugar al desarrollo de la
biopsia líquida, técnica que se basa en su evaluación
mediante pruebas de PCR y secuenciación profunda
(143-147). Esta metodología además de sus ventajas
por no ser invasiva, permite efectuar seguimiento a largo plazo de los
pacientes, evaluando información genómica, transcriptómica y
epigenetica relacionada con
la heterogeneidad tumoral, para brindar a las pacientes
alternativas terapéuticas cuando se detecten nuevas alteraciones
moleculares que provean a los tumores de ventajas proliferativas, de
escape de la respuesta inmune o
asociadas a resistencia los tratamientos (141, 142, 148).
En la actualidad la biopsia líquida ya se utiliza rutinariamente en el
estudio de pacientes con tumores metastásicos de seno (149, 150),
pulmón (151, 152), colon (153,
154), páncreas (155), melanomas (156) entre otros y sus
indicaciones ya han empezado a ser reguladas (157).
En el futuro, incluso se plantea la posibilidad de que la
biopsia líquida haga parte del abordaje diagnóstico inicial de
pacientes con cáncer junto con las biopsias de
tejidos, e inclusive reemplazándolas en algunas situaciones como
tumores de difícil acceso (142, 158, 159).
Referencias
1. Coons AH, Creech HJ, Jones RN.
Immunological Properties of an Antibody Containing a Fluorescent Group.
Exper Biol Med. 1941;47(2):200-2.
2. Taylor CR. Immunoenzyme techniques
and their
application to diagnostic studies. Ann N Y Acad Sci.
1983;420:115-26.
3. Huang SN, Minassian H, More JD.
Application of immunofluorescent staining on paraffin sections improved
by
trypsin digestion. Lab Invest. 1976;35(4):383-90.
4. Nakane PK, Pierce GB, Jr.
Enzyme-labeled antibodies
for the light and electron microscopic localization of tissue antigens.
J Cell Biol. 1967;33(2):307-18.
5. Mason TE, Phifer RF, Spicer SS,
Swallow RA, Dreskin RB. An immunoglobulin-enzyme bridge method
for localizing tissue antigens. J Histochem Cytochem.
1969;17(9):563-9.
6. Avrameas S. Enzyme markers: their
linkage with proteins and use in immuno-histochemistry. Histochem J.
1972;4(4):321-30.
7. Taylor CR, Burns J. The
demonstration of plasma cells
and other immunoglobulin-containing cells in formalinfixed,
paraffin-embedded tissues using peroxidase-labelled antibody. J Clin
Pathol. 1974;27(1):14-20.
8. Pinkus GS. Diagnostic
immunocytochemistry of paraffinembedded tissues. Hum Pathol.
1982;13(5):411-5.
9. Burns J. Background staining and
sensitivity of the unlabelled antibody-enzyme (PAP) method. Comparison
with the peroxidase labelled antibody sandwich method
using formalin fixed paraffin embedded material. Histochemistry.
1975;43(3):291-4.
10. Taylor CR. An exaltation of
experts: concerted efforts
in the standardization of immunohistochemistry. Hum
Pathol. 1994;25(1):2-11.
11. Burry RW. Specificity controls
for immunocytochemical
methods. J Histochem Cytochem. 2000;48(2):163-6.
12. Torlakovic EE, Nielsen S, Francis
G, Garratt J, Gilks B,
Goldsmith JD et al. Standardization of positive controls
in diagnostic immunohistochemistry: recommendations
from the International Ad Hoc Expert Committee. Appl
Immunohistochem Mol Morphol. 2015;23(1):1-18.
13. Torlakovic EE, Francis G, Garratt
J, Gilks B, Hyjek E,
Ibrahim M et al. Standardization of negative controls in
diagnostic immunohistochemistry: recommendations
from the international ad hoc expert panel. Appl Immunohistochem Mol
Morphol. 2014;22(4):241-52.
14. Mason JT, O'Leary TJ. Effects of
formaldehyde fixation
on protein secondary structure: a calorimetric and infrared
spectroscopic investigation. J Histochem Cytochem.
1991;39(2):225-9.
15. Werner M, Chott A, Fabiano A,
Battifora H. Effect of formalin tissue fixation and processing on
immunohistochemistry. Am J Surg Pathol. 2000;24(7):1016-9.
16. Leong AS, Gilham PN. The effects
of progressive formaldehyde fixation on the preservation of tissue
antigens. Pathology. 1989;21(4):266-8.
17. Vincek V, Nassiri M, Nadji M,
Morales AR. A Tissue
Fixative that Protects Macromolecules (DNA, RNA, and
Protein) and Histomorphology in Clinical Samples. Lab
Invest. 2003;83(10):1427-35.
18. Wester K, Asplund A, Bäckvall H,
Micke P, Derveniece A,
Hartmane I et al. Zinc-based fixative improves preservation of genomic
DNA and proteins in histoprocessing of
human tissues. Lab Invest. 2003;83(6):889-99.
19. Shibutani M, Uneyama C, Miyazaki
K, Toyoda K, Hirose
M. Methacarn Fixation: A Novel Tool for Analysis of GeneExpressions in
Paraffin-Embedded Tissue Specimens.
Lab Invest. 2000;80(2):199-208.
20. Prentø P, Lyon H. Commercial
formalin substitutes for
histopathology. Biotech Histochem. 1997;72(5):273-82.
21. Sternberger LA, Hardy PH, Jr.,
Cuculis JJ, Meyer HG.
The unlabeled antibody enzyme method of immunohistochemistry:
preparation and properties of soluble
antigen-antibody complex (horseradish peroxidaseantihorseradish
peroxidase) and its use in identification
of spirochetes. J Histochem Cytochem. 1970;18(5):315-
33.
22. DeLellis RA, Sternberger LA, Mann
RB, Banks PM,
Nakane PK. Immunoperoxidase technics in diagnostic
pathology. Report of a workshop sponsored by the National Cancer
Institute. Am J Clin Pathol. 1979;71(5):483-8.
23. Hsu SM, Raine L, Fanger H. Use of
avidin-biotin-peroxidase complex (ABC) in immunoperoxidase techniques:
a
comparison between ABC and unlabeled antibody (PAP)
procedures. J Histochem Cytochem. 1981;29(4):577-80.
24. Levsky JM, Singer RH.
Fluorescence in situ hybridization: past, present and future. J Cell
Sci. 2003;116(Pt
14):2833-8.
25. Streefkerk JG. Inhibition of
erythrocyte pseudoperoxidase activity by treatment with hydrogen
peroxide following
methanol. J Histochem Cytochem. 1972;20(10):829-31.
26. Weir E, Pretlow T, Pitts A,
Williams EE. Destruction of
endogenous peroxidase activity in order to locate cellular antigens by
peroxidase-labeled antibodies. J Histochem Cytochem. 1974;22:51 - 4.
27. Andrew SM, Jasani B. An improved
method for the inhibition of endogenous peroxidase non-deleterious to
lymphocyte surface markers. Application to immunoperoxidase studies on
eosinophil-rich tissue preparations.
Histochem J. 1987;19(8):426-30.
28. Cordell JL, Falini B, Erber WN,
Ghosh AK, Abdulaziz Z,
MacDonald S et al. Immunoenzymatic labeling of monoclonal antibodies
using immune complexes of alkaline
phosphatase and monoclonal anti-alkaline phosphatase (APAAP complexes).
J Histochem Cytochem.
1984;32(2):219-29.
29. Wagner L, Worman CP.
Color-contrast staining of two
different lymphocyte subpopulations: a two-color modification of
alkaline phosphatase monoclonal anti-alkaline phosphatase complex
technique. Stain Technol.
1988;63(3):129-36.
30. Kuhlmann WD, Peschke P. Glucose
oxidase as label in
histological immunoassays with enzyme-amplification
in a two-step technique: coimmobilized horseradish peroxidase as
secondary system enzyme for chromogen
oxidation. Histochemistry. 1986;85(1):13-7.
31. Mason DY, Sammons R. Alkaline
phosphatase and peroxidase for double immunoenzymatic labelling of
cellular constituents. J Clin Pathol. 1978;31(5):454-60.
32. Faulk WP, Taylor GM. An
immunocolloid method
for the electron microscope. Immunochemistry.
1971;8(11):1081-3.
33. Krenács T, Lászik Z, Dobó E.
Application of immunogold-silver staining and immunoenzymatic methods
in
multiple labelling of human pancreatic Langerhans islet
cells. Acta Histochem. 1989;85(1):79-85.
34. ADL. DP, Mukdsi J, JP. P,
Gutiérrez S, AA. Q, CA M et
al. Immunoelectron Microscopy: A Reliable Tool for the
Analysis of Cellular Processes. En: IntechOpen, editor.
Applications of Immunohistochemistry; 2011.
35. Park C-H, Kim H, Chang B-J, Lee
S, Chang B, Bae C-S
et al. Overview of Immunoelectron Microscopy. Appl Microsc.
2018;48:87-95.
36. Fraenkel-Conrat H, Olcott HS. The
reaction of formaldehyde with proteins; cross-linking between amino and
primary amide or guanidyl groups. J Am Chem Soc.
1948;70(8):2673-84.
37. Fraenkel-Conrat H, Olcott HS.
Reaction of formaldehyde with proteins; cross-linking of amino groups
with phenol, imidazole, or indole groups. J Biol Chem.
1948;174(3):827-43.
38. Huang SN. Immunohistochemical
demonstration of hepatitis B core and surface antigens in paraffin
sections.
Lab Invest. 1975;33(1):88-95.
39. Battifora H, Kopinski M. The
influence of protease digestion and duration of fixation on the
immunostaining of
keratins. A comparison of formalin and ethanol fixation. J
Histochem Cytochem. 1986;34(8):1095-100.
40. Köhler G, Milstein C. Continuous
cultures of fused cells
secreting antibody of predefined specificity. Nature.
1975;256(5517):495-7.
41. Taylor CR. The total test
approach to standardization of immunohistochemistry. Arch Pathol Lab
Med.
2000;124(7):945-51.
42. Swaab DF, Pool CW, Van Leeuwen
FW. Can specificity
ever be proved in immunocytochemical staining. J Histochem Cytochem.
43. Fitzgibbons PL, Bradley LA,
Fatheree LA, Alsabeh R,
Fulton RS, Goldsmith JD et al. Principles of Analytic
Validation of Immunohistochemical Assays: Guideline
From the College of American Pathologists Pathology
and Laboratory Quality Center. Arch Pathol Lab Med.
2014;138(11):1432-43.
44. Fetsch PA, Abati A. Overview of
the Clinical Immunohistochemistry Laboratory: Regulations and
Troubleshooting Guidelines. En: Javois LC, editor. Immunocytochemical
Methods and Protocols. Totowa, NJ: Humana
Press; 1999.
45. Bass BP, Engel KB, Greytak SR,
Moore HM. A review
of preanalytical factors affecting molecular, protein, and
morphological analysis of formalin-fixed, paraffin-embedded (FFPE)
tissue: how well do you know your FFPE specimen? Arch Pathol Lab Med.
2014;138(11):1520-
30.
46. Wester K, Wahlund E, Sundström C,
Ranefall P, Bengtsson E, Russell PJ et al. Paraffin section storage and
immunohistochemistry. Effects of time, temperature, fixation, and
retrieval protocol with emphasis on p53 protein
and MIB1 antigen. Appl Immunohistochem Mol Morphol.
2000;8(1):61-70.
47. Xie R, Chung JY, Ylaya K,
Williams RL, Guerrero N,
Nakatsuka N et al. Factors influencing the degradation
of archival formalin-fixed paraffin-embedded tissue sections. J
Histochem Cytochem. 2011;59(4):356-65.
48. Leong A, Millos J. An Assessment
of the Efficacy
of the Microwave Antigen-Retrieval Procedure on a
Range of Tissue Antigens. Appl Immunohistochem.
1993;1(4):267-74.
49. Shi SR, Imam SA, Young L, Cote
RJ, Taylor CR. Antigen retrieval immunohistochemistry under the
influence
of pH using monoclonal antibodies. J Histochem Cytochem.
1995;43(2):193-201.
50. Shi S, Cote R, Taylor C.
Standardization and further
development of antigen retrieval immunohistochemistry: Strategies and
future goals. J Histotechnol.
1999;22:177-92.
51. Grabau D, Nielsen O, Hansen SR,
Nielsen MM, nkholm
A-VL, Knoop A et al., editores. Influence of Storage
Temperature and High-Temperature Antigen Retrieval
Buffers on Results of Immunohistochemical Staining in
Sections Stored for Long Periods; 1998.
52. Taylor CR, Shi SR, Chen C, Young
L, Yang C, Cote RJ.
Comparative study of antigen retrieval heating methods:
microwave, microwave and pressure cooker, autoclave,
and steamer. Biotech Histochem. 1996;71(5):263-70.
53. Heras A, Roach CM, Key ME.
Enhanced Polymer Detection System for Immunohistochemistry. Lab Invest.
1995;72(1):A165-A.
54. Vyberg M, Nielsen SR, editores.
Dextran Polymer Conjugate Two-Step Visualization System for
Immunohistochemistry: A Comparison of EnVision+ With Two ThreeStep
Avidin-Biotin Techniques; 1998.
55. Sabattini E, Bisgaard K, Ascani
S, Poggi S, Piccioli
M, Ceccarelli C et al. The EnVision++ system: a new
immunohistochemical method for diagnostics and research. Critical
comparison with the APAAP, ChemMate, CSA, LABC, and SABC techniques. J
Clin Pathol.
1998;51(7):506-11.
56. Yaziji H, Taylor CR, Goldstein
NS, Dabbs DJ, Hammond
EH, Hewlett B et al. Consensus recommendations on
estrogen receptor testing in breast cancer by immunohistochemistry.
Appl Immunohistochem Mol Morphol.
2008;16(6):513-20.
57. Harvey JM, Clark GM, Osborne CK,
Allred DC. Estrogen
receptor status by immunohistochemistry is superior to
the ligand-binding assay for predicting response to adjuvant endocrine
therapy in breast cancer. J Clin Oncol.
1999;17(5):1474-81.
58. Rhodes A, Jasani B, Balaton AJ,
Barnes DM, Miller KD.
Frequency of oestrogen and progesterone receptor positivity by
immunohistochemical analysis in 7016 breast
carcinomas: correlation with patient age, assay sensitivity, threshold
value, and mammographic screening. J
Clin Pathol. 2000;53(9):688-96.
59. Yaziji H, Taylor CR. Begin at the
beginning, with the tissue! The key message underlying the ASCO/CAP
Taskforce Guideline Recommendations for HER2 testing.
Applied immunohistochemistry & molecular morphology : AIMM.
2007;15(3):239-41.
60. Wolff AC, Hammond ME, Schwartz
JN, Hagerty KL,
Allred DC, Cote RJ et al. American Society of Clinical
Oncology/College of American Pathologists guideline
recommendations for human epidermal growth factor receptor 2 testing in
breast cancer. J Clin Oncol.
2007;25(1):118-45.
61. Taylor CR, Levenson RM.
Quantification of immunohistochemistry--issues concerning methods,
utility
and semiquantitative assessment II. Histopathology.
2006;49(4):411-24.
62. Taylor CR. Quantifiable internal
reference standards
for immunohistochemistry: the measurement of quantity by weight. Appl
Immunohistochem Mol Morphol.
2006;14(3):253-9.
63. Herman GE, Elfont EA, Floyd AD.
Overview of automated immunostainers. Methods Mol Biol. 1994;34:383-
403.
64. Le Neel T, Moreau A, Laboisse C,
Truchaud A. Comparative evaluation of automated systems in
immunohistochemistry. Clin Chim Acta. 1998;278(2):185-92.
65. Moreau A, Néel TL, Joubert M,
Truchaud
A, Laboisse C.
Approach to automation in immunohistochemistry.Clin
Chim Acta. 1998;278 2:177-84.
66. Hendry S, Salgado R, Gevaert T,
Russell PA, John T,
Thapa B et al. Assessing Tumor-Infiltrating Lymphocytes
in Solid Tumors: A Practical Review for Pathologists and
Proposal for a Standardized Method from the International
Immuno-Oncology Biomarkers Working Group: Part
2: TILs in Melanoma, Gastrointestinal Tract Carcinomas,
Non-Small Cell Lung Carcinoma and Mesothelioma,
Endometrial and Ovarian Carcinomas, Squamous Cell
Carcinoma of the Head and Neck, Genitourinary Carcinomas, and Primary
Brain Tumors. Adv Anat Pathol.
2017;24(6):311-35.
67. Hendry S, Salgado R, Gevaert T,
Russell PA, John T,
Thapa B et al. Assessing Tumor-infiltrating Lymphocytes
in Solid Tumors: A Practical Review for Pathologists and
Proposal for a Standardized Method From the International
Immunooncology Biomarkers Working Group: Part 1: Assessing the Host
Immune Response, TILs in Invasive Breast Carcinoma and Ductal Carcinoma
In Situ, Metastatic Tumor Deposits and Areas for Further Research.
Adv Anat Pathol. 2017;24(5):235-51.
68. Gorris MAJ, Halilovic A, Rabold
K, van Duffelen A, Wickramasinghe IN, Verweij D et al. Eight-Color
Multiplex
Immunohistochemistry for Simultaneous Detection of
Multiple Immune Checkpoint Molecules within the Tumor
Microenvironment. J Immunol. 2018;200(1):347-54.
69. Klauschen F, Müller KR, Binder A,
Bockmayr M, Hägele
M, Seegerer P, et al. Scoring of tumor-infiltrating lymphocytes: From
visual estimation to machine learning.
Semin Cancer Biol. 2018;52(Pt 2):151-7.
70. H. R. Tumors of the hematopoietic
system. In: Atlas of
Tumor Pathology. Washington D.C: Armed Institute of
Pathology 1966.
71. Lukes RJ, Collins RD. Immunologic
characterization
of human malignant lymphomas. Cancer. 1974;34(4
Suppl):suppl:1488-503.
72. National Cancer Institute
sponsored study of classifications of non-Hodgkin's lymphomas: summary
and
description of a working formulation for clinical usage.
The Non-Hodgkin's Lymphoma Pathologic Classification
Project. Cancer. 1982;49(10):2112-35.
73. Bernard A, Boumsell L. The
clusters of differentiation
(CD) defined by the First International Workshop on Human Leucocyte
Differentiation Antigens. Hum Immunol.
1984;11(1):1-10.
74. Subcommittee I-WN. Nomenclature
for clusters of differentiation (CD) of antigens defined on human
leukocyte
populations. IUIS-WHO Nomenclature Subcommittee.
Bull World Health Organ. 1984;62(5):809-15.
75. C.R. T, R.J. C. Tumors of unknown
origin. En: C.R T, editor. Immunomicroscopy A diagnostic tool for the
Surgical
Pathologist Major Problems in Pathology. 3 ed: Saunders; 2006.
76. D.J. D. Immunohistology of
metastatic carcinoma of unknown primary site. En: R. B, D.J. D,
editores. Diagnostic
immunohistochemistry. Theranostic and Genomic Applications. 5 ed:
Elsevier; 2019.
77. Hofman V, Lassalle S, Bence C,
Long-Mira E, NahonEstève S, Heeke S et al. Any Place for
Immunohistochemistry within the Predictive Biomarkers of Treatment in
Lung Cancer Patients? Cancers (Basel). 2018;10(3):70.
78. Gu J, Taylor CR. Practicing
pathology in the era of big
data and personalized medicine. Applied immunohistochemistry &
molecular morphology: AIMM. 2014;22(1):1-
9.
79. Taylor CR. Predictive biomarkers
and companion diagnostics. The future of immunohistochemistry: "in situ
proteomics," or just a "stain"? Applied immunohistochemistry &
molecular morphology : AIMM. 2014;22(8):555-
61.
80. Yuan J, Hegde PS, Clynes R,
Foukas PG, Harari A,
Kleen TO et al. Novel technologies and emerging biomarkers for
personalized cancer immunotherapy. J Immunother Cancer. 2016;4(1):3.
81. Fisher B, Redmond C, Brown A,
Wickerham DL, Wolmark N, Allegra J et al. Influence of tumor estrogen
and
progesterone receptor levels on the response to tamoxifen and
chemotherapy in primary breast cancer. J Clin
Oncol. 1983;1(4):227-41.
82. Lerner LJ, Jordan VC. Development
of antiestrogens
and their use in breast cancer: eighth Cain memorial
award lecture. Cancer Res. 1990;50(14):4177-89.
83. Jordan VC. Tamoxifen: a personal
retrospective. Lancet
Oncol. 2000;1(1):43-9.
84. Druker BJ, Tamura S, Buchdunger
E, Ohno S, Segal
GM, Fanning S et al. Effects of a selective inhibitor of
the Abl tyrosine kinase on the growth of Bcr-Abl positive
cells. Nat Med. 1996;2(5):561-6.
85. Goldman JM, Druker BJ. Chronic
myeloid leukemia: current treatment options. Blood. 2001;98(7):2039-42.
86. Slamon DJ, Clark GM, Wong SG,
Levin WJ, Ullrich A,
McGuire WL. Human breast cancer: correlation of relapse and survival
with amplification of the HER-2/neu
oncogene. Science. 1987;235(4785):177-82.
87. Hanna WM, Hammond E, Taylor CR,
Dabbs DJ, Penault-Llorca F, Bloom KJ et al. Re: High concordance
between immunohistochemistry and fluorescence in situ
hybridization testing for HER2 status in breast cancer
requires a normalized IHC scoring system. Mod Pathol.
2008;21(10):1278-80; author reply 80-1.
88. Slamon DJ, Leyland-Jones B, Shak
S, Fuchs H, Paton V, Bajamonde A et al. Use of chemotherapy plus
a monoclonal antibody against HER2 for metastatic
breast cancer that overexpresses HER2. N Engl J Med.
2001;344(11):783-92.
89. Foran JM, Cunningham D, Coiffier
B, Solal-Celigny P,
Reyes F, Ghielmini M et al. Treatment of mantle-cell lymphoma with
Rituximab (chimeric monoclonal anti-CD20
antibody): analysis of factors associated with response.
Ann Oncol. 2000;11 Suppl 1:117-21.
90. In Vitro Diagnostics [Internet].
U.S. Food and Drug
Administration.2016 [consultado 09 noviembre 2020].
Disponible en:
http://www.fda.gov/Medical-Devices/Products-and-Medical-Procedures/In-Vitro-Diagnostics/ucm301431.htm. .
91. Matos LLd, Trufelli DC, de Matos
MGL, da Silva Pinhal MA. Immunohistochemistry as an important tool in
biomarkers detection and clinical practice. Biomark Insights.
2010;5:9-20.
92. Nowell PC. The minute chromosome
(Phl) in chronic
granulocytic leukemia. Blut. 1962;8:65-6.
93. J. R. Chromosomes in leukemia and
lymphoma. Seminars in Hematology. 1978;15(3):301-19.
94. Watson JD, Crick FHC. Genetical
Implications of
the Structure of Deoxyribonucleic Acid. Nature.
1953;171(4361):964-7.
95. Watson JD, Crick FHC. Molecular
Structure of Nucleic
Acids: A Structure for Deoxyribose Nucleic Acid. Nature.
1953;171(4356):737-8.
96. van der Ploeg M. Cytochemical
nucleic acid research during the twentieth century. Eur J Histochem.
2000;44(1):7-42.
97. Smeets DF. Historical prospective
of human cytogenetics: from microscope to microarray. Clin Biochem.
2004;37(6):439-46.
98. Speicher MR, Carter NP. The new
cytogenetics: blurring
the boundaries with molecular biology. Nat Rev Genet.
2005;6(10):782-92.
99. Penault-Llorca F, Bilous M,
Dowsett M, Hanna W, Osamura RY, Rüschoff J, et al. Emerging
technologies
for assessing HER2 amplification. Am J Clin Pathol.
2009;132(4):539-48.
100. Gall JG. The origin of in situ
hybridization - A personal
history. Methods. 2016;98:4-9.
101. Gall JG, Pardue ML. Formation
and detection of RNADNA hybrid molecules in cytological preparations.
Proc
Natl Acad Sci U S A. 1969;63(2):378-83.
102. John HA, Birnstiel ML, Jones KW.
RNA-DNA hybrids at
the cytological level. Nature. 1969;223(5206):582-7.
103. Buongiorno-Nardelli M, Amaldi F.
Autoradiographic detection of molecular hybrids between RNA and DNA in
tissue sections. Nature. 1970;225(5236):946-8.
104. Bauman JG, Wiegant J, Borst P,
van Duijn P. A new
method for fluorescence microscopical localization of
specific DNA sequences by in situ hybridization of fluorochromelabelled
RNA. Exp Cell Res. 1980;128(2):485-
90.
105. Wilcox JN. Fundamental
principles of in situ hybridization. J Histochem Cytochem.
1993;41(12):1725-33.
106. Jensen E. Technical review: In
situ hybridization. Anat
Rec (Hoboken). 2014;297(8):1349-53.
107. Tanner M, Gancberg D, Di Leo A,
Larsimont D, Rouas
G, Piccart MJ et al. Chromogenic in situ hybridization: a
practical alternative for fluorescence in situ hybridization
to detect HER-2/neu oncogene amplification in archival
breast cancer samples. Am J Pathol. 2000;157(5):1467-
72.
108. Park K, Han S, Kim J-Y, Kim H-J,
Kwon JE, Gwak G.
Silver-Enhanced In Situ Hybridization as an Alternative
to Fluorescence In Situ Hybridization for Assaying HER2
Amplification in Clinical Breast Cancer. J Breast Cancer.
2011;14(4):276-82.
109. Pedersen M, Rasmussen BB. The
correlation between dual-color chromogenic in situ hybridization and
fluorescence in situ hybridization in assessing HER2
gene amplification in breast cancer. Diagn Mol Pathol.
2009;18(2):96-102.
110. Gulley ML, Glaser SL, Craig FE,
Borowitz M, Mann RB,
Shema SJ et al. Guidelines for interpreting EBER in situ
hybridization and LMP1 immunohistochemical tests for
detecting Epstein-Barr virus in Hodgkin lymphoma. Am J
Clin Pathol. 2002;117(2):259-67.
111. Cook JR. Paraffin section
interphase fluorescence in situ
hybridization in the diagnosis and classification of nonhodgkin
lymphomas. Diagn Mol Pathol. 2004;13(4):197-
206.
112. Liu Y, Barta SK. Diffuse large
B-cell lymphoma: 2019 update on diagnosis, risk stratification, and
treatment. Am
J Hematol. 2019;94(5):604-16.
113. Ting C-Y, Chang K-M, Kuan J-W,
Sathar J, Chew L-P,
Wong O-LJ et al. Clinical Significance of BCL2, C-MYC,
and BCL6 Genetic Abnormalities, Epstein-Barr Virus
Infection, CD5 Protein Expression, Germinal Center B
Cell/Non-Germinal Center B-Cell Subtypes, Co-expression of MYC/BCL2
Proteins and Co-expression of MYC/
BCL2/BCL6 Proteins in Diffuse Large B-Cell Lymphoma:
A Clinical and Pathological Correlation Study of 120 Patients. Int J
Med Sci. 2019;16(4):556-66
114. Ma Z, Niu J, Cao Y, Pang X, Cui
W, Zhang W et al. Clinical significance of 'double-hit' and
'double-expression'
lymphomas. J Clin Pathol. 2020;73(3):126-38.
115. Thunnissen E, Bubendorf L,
Dietel M, Elmberger G, Kerr
K, Lopez-Rios F, et al. EML4-ALK testing in non-small
cell carcinomas of the lung: a review with recommendations. Virchows
Arch. 2012;461(3):245-57.
116. Schildhaus HU, Deml KF, Schmitz
K, Meiboom M, Binot E, Hauke S, et al. Chromogenic in situ
hybridization is a reliable assay for detection of ALK rearrangements
in adenocarcinomas of the lung. Mod Pathol.
2013;26(11):1468-77.
117. Luk PP, Selinger CI, Cooper WA,
Mahar A, Palme CE,
O'Toole SA, et al. Clinical Utility of In Situ Hybridization Assays in
Head and Neck Neoplasms. Head Neck
Pathol. 2019;13(3):397-414.
118. Ferrara G, De Vanna AC.
Fluorescence In Situ Hybridization for Melanoma Diagnosis: A Review and
a Reappraisal. Am J Dermatopathol. 2016;38(4):253-69.
119. Tanas MR, Goldblum JR.
Fluorescence in situ hybridization in the diagnosis of soft tissue
neoplasms: a review.
Adv Anat Pathol. 2009;16(6):383-91.
120. Nitta H, Hauss-Wegrzyniak B,
Lehrkamp M, Murillo AE,
Gaire F, Farrell M et al. Development of automated brightfield double
in situ hybridization (BDISH) application for
HER2 gene and chromosome 17 centromere (CEN 17)
for breast carcinomas and an assay performance comparison to manual
dual color HER2 fluorescence in situ
hybridization (FISH). Diagn Pathol. 2008;3:41121.
121. Sanger F, Coulson AR. A rapid
method for determining
sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol.
1975;94(3):441-8.
122. Heather JM, Chain B. The
sequence of sequencers: The
history of sequencing DNA. Genomics. 2016;107(1):1-8.
123. Mullis K, Faloona F, Scharf S,
Saiki R, Horn G, Erlich H.
Specific enzymatic amplification of DNA in vitro: the polymerase chain
reaction. Cold Spring Harb Symp Quant
Biol. 1986;51(Pt 1):263-73.
124. Mullis KB, Faloona FA. Specific
synthesis of DNA in vitro
via a polymerase-catalyzed chain reaction. Methods Enzymol.
1987;155:335-50.
125. Mullis KB. The unusual origin of
the polymerase chain
reaction. Sci Am. 1990;262(4):56-61, 4-5.
126. Nathwani BN, Sasu SJ, Ahsanuddin
AN, Hernandez AM,
Drachenberg MR. The critical role of histology in an era
of genomics and proteomics: a commentary and reflection. Adv Anat
Pathol. 2007;14(6):375-400.
127. Eltoum IA, Siegal GP, Frost AR.
Microdissection of histologic sections: past, present, and future. Adv
Anat
Pathol. 2002;9(5):316-22.
128. Hunt JL, Finkelstein SD.
Microdissection techniques for
molecular testing in surgical pathology. Arch Pathol Lab
Med. 2004;128(12):1372-8.
129. Espina V, Heiby M, Pierobon M,
Liotta LA. Laser capture microdissection technology. Expert Rev Mol
Diagn.
2007;7(5):647-57.
130. Going J. Histological
microdissection in diagnostic and
investigative pathology. Diagnostic Histopathology.
2010;16:43-8.
131. Goelz SE, Hamilton SR,
Vogelstein B. Purification
of DNA from formaldehyde fixed and paraffin embedded human tissue.
Biochem Biophys Res Commun.
1985;130(1):118-26.
132. Bianchi AB, Navone NM, Conti CJ.
Detection of loss of
heterozygosity in formalin-fixed paraffin-embedded tumor specimens by
the polymerase chain reaction. Am J
Pathol. 1991;138(2):279-84.
133. Going JJ, Lamb RF. Practical
histological microdissection for PCR analysis. J Pathol.
1996;179(1):121-4.
134. Emmert-Buck MR, Bonner RF, Smith
PD, Chuaqui RF,
Zhuang Z, Goldstein SR et al. Laser capture microdissection. Science.
1996;274(5289):998-1001.
135. Gross A, Schoendube J,
Zimmermann S, Steeb M, Zengerle R, Koltay P. Technologies for
Single-Cell Isolation.
Int J Mol Sci. 2015;16(8):16897-919.
136. d'Amore F, Stribley JA, Ohno T,
Wu G, Wickert RS, Delabie J et al. Molecular studies on single cells
harvested
by micromanipulation from archival tissue sections previously stained
by immunohistochemistry or nonisotopic
in situ hybridization. Lab Invest. 1997;76(2):219-24.
137. Fend F, Raffeld M. Laser capture
microdissection in
pathology. J Clin Pathol. 2000;53(9):666-72.
138. Pantel K, Alix-Panabières C.
Circulating tumour cells in
cancer patients: challenges and perspectives. Trends
Mol Med. 2010;16(9):398-406.
139. Cristofanilli M, Budd GT, Ellis
MJ, Stopeck A, Matera J,
Miller MC et al. Circulating tumor cells, disease progression, and
survival in metastatic breast cancer. N Engl J
Med. 2004;351(8):781-91.
140. Francis G, Stein S. Circulating
Cell-Free Tumour
DNA in the Management of Cancer. Int J Mol Sci.
2015;16(6):14122-42.
141. Finotti A, Allegretti M,
Gasparello J, Giacomini P, Spandidos DA, Spoto G et al. Liquid biopsy
and PCR-free
ultrasensitive detection systems in oncology (Review).
Int J Oncol. 2018;53(4):1395-434.
142. Fernández-Lázaro D, Hernández
JLG, García AC, Castillo ACD, Hueso MV, Cruz-Hernández JJ. Clinical
Perspective and Translational Oncology of Liquid Biopsy.
Diagnostics (Basel).2020;10(7):443.
143. Cappelletti V, Appierto V,
Tiberio P, Fina E, Callari M,
Daidone MG. Circulating Biomarkers for Prediction
of Treatment Response. J Natl Cancer Inst Monogr.
2015;2015(51):60-3.
144. von Bubnoff N. Liquid Biopsy:
Approaches to Dynamic
Genotyping in Cancer. Oncol Res Treat. 2017;40(7-
8):409-16.
145. Arneth B. Update on the types
and usage of liquid biopsies in the clinical setting: a systematic
review. BMC
Cancer. 2018;18(1):527.
146. Bennett CW, Berchem G, Kim YJ,
El-Khoury V. Cell-free
DNA and next-generation sequencing in the service of
personalized medicine for lung cancer. Oncotarget.
2016;7(43):71013-35.
147. Cristiano S, Leal A, Phallen J,
Fiksel J, Adleff V, Bruhm
DC, et al. Genome-wide cell-free DNA fragmentation in
patients with cancer. Nature. 2019;570(7761):385-9.
148. Wang J, Chang S, Li G, Sun Y.
Application of liquid biopsy in precision medicine: opportunities and
challenges.
Front Med. 2017;11(4):522-7.
149. Alimirzaie S, Bagherzadeh M,
Akbari MR. Liquid biopsy
in breast cancer: A comprehensive review. Clin Genet.
2019;95(6):643-60.
150. Koch C, Kuske A, Joosse SA,
Yigit G, Sflomos G, Thaler S et al. Characterization of circulating
breast cancer
cells with tumorigenic and metastatic capacity. EMBO
Mol Med. 2020;12(9):e11908.
151. Johann DJ, Jr., Steliga M, Shin
IJ, Yoon D, Arnaoutakis
K, Hutchins L et al. Liquid biopsy and its role in an advanced clinical
trial for lung cancer. Exp Biol Med (Maywood). 2018;243(3):262-71.
152. Couraud S, Vaca-Paniagua F,
Villar S, Oliver J, Schuster
T, Blanché H et al. Noninvasive diagnosis of actionable
mutations by deep sequencing of circulating free DNA
in lung cancer from never-smokers: a proof-of-concept study from
BioCAST/IFCT-1002. Clin Cancer Res.
2014;20(17):4613-24.
153. Wills B, Gorse E, Lee V. Role of
liquid biopsies in colorectal cancer. Curr Probl Cancer.
2018;42(6):593-600.
154. Marcuello M, Vymetalkova V,
Neves RPL, Duran-Sanchon S, Vedeld HM, Tham E et al. Circulating
biomarkers for early detection and clinical management of colorectal
cancer. Mol Aspects Med. 2019;69:107-22.
155. Gao Y, Zhu Y, Yuan Z.
Circulating Tumor Cells and Circulating Tumor DNA Provide New Insights
into Pancreatic Cancer. Int J Med Sci. 2016;13(12):902-13.
156. Gray ES, Reid AL, Bowyer S,
Calapre L, Siew K, Pearce R, et al. Circulating Melanoma Cell
Subpopulations:
Their Heterogeneity and Differential Responses to
Treatment. J Invest Dermatol. 2015;135(8):2040-8.
157. Strotman LN, Millner LM, Valdes
R, Jr., Linder MW. Liquid Biopsies in Oncology and the Current
Regulatory
Landscape. Mol Diagn Ther. 2016;20(5):429-36.
158. Siravegna G, Mussolin B, Venesio
T, Marsoni S, Seoane
J, Dive C et al. How liquid biopsies can change clinical
practice in oncology. Ann Oncol. 2019;30(10):1580-90.
159. Rossi G, Ignatiadis M. Promises
and Pitfalls of Using
Liquid Biopsy for Precision Medicine. Cancer Res.
2019;79(11):2798-804.
Recibido:
Noviembre 13, 2020
Aprobado: Diciembre 15, 2020
Correspondencia:
María del Pilar Archila-Gomez
archilapilar@yahoo.com







