Resumen
Objetivo: Determinar el impacto en la sobrevida que presentan las mutaciones de sensibilidad y de amplificación de EGFR en pacientes con adenocarcinoma de pulmón (ADP) avanzado, tratados con Inhibidores de Tirosina Quinasa (TKI). Métodos: Se realizó amplificación de EGFR en 72 pacientes con ADP que presentan mutaciones activadoras de EGFR y que recibieron Erlotinib como tratamiento. Se analizó la relación entre el estado mutacional de EGFR y el perfil del número de copias, con la tasa de respuesta, la supervivencia general (SG) y la supervivencia libre de progresión (SLP). Resultados: La edad media fue de 62 años (r, 20-87 años), 53 pacientes fueron mujeres (73%) y 89% tuvieron mutaciones comunes. Veintidós (30,6%) muestras con mutaciones activantes de EGFR fueron identificadas mediante la amplificación de EGFR. Esto fue más frecuente en pacientes con deleción del exón 19 (p = 0,05) y con un mejor estado funcional (p = 0,01). Los pacientes con amplificación de EGFR tuvieron una SLP mayor en comparación con los pacientes sin amplificación de EGFR [(25,2 meses, IC 95% 22,0-38,5) vs. (12,4 meses, IC 95% 5,3-19,5); p = 0,002]; también presentaron una supervivencia global (SG) mejor [(37,8 meses, IC 95% 30,9-44,7) vs. (27,1 meses, IC 95% 12,8-41,3); p = 0,009]. La amplificación de EGFR influyó de forma significativa en la respuesta al tratamiento con Erlotinib (p = 0,0001). Conclusión: En la población estudiada, la amplificación de EGFR y las mutaciones sensibilizantes son biomarcadores asociados a una mejor supervivencia global, supervivencia libre de progresión y respuesta a la terapia con EGFR-TKI en pacientes con ADP avanzado.
Palabras clave: adenocarcinoma de pulmón; factor de crecimiento epidérmico; Erlotinib; supervivencia.

1 MD. Fundación para la
Investigación Clínica y Molecular Aplicada del Cáncer, FICMAC. Bogotá,
Colombia.
2 MD. Fundación para la Investigación Clínica y Molecular Aplicada
del Cáncer, FICMAC. Bogotá, Colombia.
3 MD. Fundación para la Investigación Clínica y Molecular Aplicada
del Cáncer, FICMAC. Bogotá, Colombia.
4 MD. MSc. PhD. Grupo de Oncología Clínica y Traslacional, Clínica
del Country. Fundación para la Investigación Clínica y Aplicada
del Cáncer, FICMAC. Grupo para la Investigación en Sistemas de
Oncología Molecular y Biología (FOX-G), Universidad el Bosque.
Bogotá, Colombia.
5 MD. MSc. Oncólogo clínico. Grupo de Oncología Clínica y
Traslacional, Instituto de Oncología, Clínica del Country. Departamento
de Oncología Clínica, Clínica Colsanitas. Bogotá, Colombia.
6 MD. Oncólogo Clínico. Fundación para la Investigación Clínica y
Aplicada del Cáncer, FICMAC. Grupo de Oncología Clínica y
Traslacional, Instituto de Oncología, Clínica del Country. Bogotá,
Colombia.
7 MD. Oncólogo Clínico. Fundación para la Investigación Clínica y
Aplicada del Cáncer, FICMAC. Grupo de Oncología Clínica y
Traslacional, Instituto de Oncología, Clínica del Country. Bogotá,
Colombia.
8 MD. Oncólogo Clínico. Fundación para la Investigación Clínica y
Aplicada del Cáncer, FICMAC. Grupo de Oncología Clínica y
Traslacional, Instituto de Oncología, Clínica del Country. Bogotá,
Colombia.
9 MD. Patóloga. Fundación para la Investigación Clínica y Aplicada
del Cáncer, FICMAC. Bogotá, Colombia.
10 Lic. Biología. MSc. Fundación para la Investigación Clínica y
Aplicada del Cáncer, FICMAC. Bogotá, Colombia.
11 Microbióloga. MSc. Fundación para la Investigación Clínica y
Aplicada del Cáncer, FICMAC. Bogotá, Colombia.
12 Bióloga. MSc. Fundación para la Investigación Clínica y Aplicada
del Cáncer, FICMAC. Bogotá, Colombia.
13 MD. MSc. Epidemióloga. Fundación para la Investigación Clínica y
Aplicada del Cáncer, FICMAC. Bogotá, Colombia.
14 MD. Hemato-Oncólogo. Grupo de Oncología Clínica, Fundación Santa
Fe de Bogotá. Bogotá, Colombia.
EGFR AMPLIFICATION AND SENSITIZING MUTATIONS CORRELATE WITH SURVIVAL FROM ERLOTINIB IN LUNG ADENOCARCINOMA PATIENTS (MUTP-CLICAP* )
RUNNING TITLE: EGFR AMPLIFICATION IN LUNG CANCER
Abstract
Background: Non-small cell lung cancer (NSCLC) has a 5-year survival of 5-16%. EGFR mutations, in most cases confer sensitivity to EGFR- TKI therapy. Nonetheless, it is still unclear why clinical outcomes vary among patients with identical EGFR mutations. The presence of EGFR amplification (EGFRamp) may play a significant role. Objective: Compare the complete (CR) and partial response (CR), overall survival (OR) and progression free survival (PFS) in Hispanic patients with lung adenocarcinoma treated with erlotinib with EGFR mutations (L58R or exon 19 deletion [Del 19]) with and without concomitant EGFRamp. Patients and Methods: 72 EGFR positive lung adenocarcinoma patients of Hispanic origin, who underwent first line treatment with erlotinib, were evaluated for EGFRamp by FISH. The clinical outcomes we analyzed according to EGFR mutations and EGFRamp status. Results: 30,6% samples had EGFRamp, more frequently present in patients with Del 19 (p=0,05). Patients with EGFRamp had a longer PFS than those without (in months) [(28,5, 95%CI 22,3- 34,6) vs. (11,0, 95%CI 8,2-16,7); p=0,002] as well as better OS [(37,8, 95%CI 30,9-44,7) vs. (27,1, 95%CI 12,8-41,3); p=0,009]. EGFRamp significantly influenced the response to erlotinib (p=0,0001). EGFRamp+/Del19 had a longer OS, 37,8 (95%CI 31,0-44,6) compared to EGFRamp+/L8585R, 27,5 (95%CI 12,4-42,5) (p<0,001) as well as a longer PFS (p=0,043). Conclusion: Among Hispanic patients, EGFRamp was present in 30% of patients with EGFR mutations. EGFR mutations and EGFRamp are associated with better OS, PFS, CR and PR to erlotinib and hence could aid in the correct selection of patients that would benefit from EGFR-TKI treatment.Key points:
- Similar to other populations, Hispanic patients with lung adenocarcinoma have EGFR mutations and EGFR amplification concomitantly in 30,6% of cases. This genetic phenomenon is significantly associated with improved outcomes, including progression-free and overall survival.
- Detection methods for EGFR mutations and concomitant EGFR amplification on patients with lung adenocarcinoma can potentially select in an accurate manner patients that will benefit from TKI therapy.
Introducción
El cáncer de pulmón no microcítico (NSCLC por sus siglas en inglés) es la causa más frecuente de mortalidad relacionada con cáncer en el mundo, con una supervivencia aproximada a 5 años del 15% (1). Se cree que las mutaciones del receptor del factor de crecimiento epidérmico (EGFR) son un evento molecular temprano en la génesis del NSCLC, particularmente en los no fumadores (2-4). Estas mutaciones ocurren aproximadamente en el 10% de los pacientes con NSCLC de etnia caucásica, y hasta 20 al 50% de los pacientes con adenocarcinoma de pulmón (ADC) en los países de Asia Oriental y América Latina (5-9).Los estudios con Gefitinib y Erlotinib
Los primeros Inhibidores de Tirosina Quinasa (TKIs) aprobados para el tratamiento del NSCLC mutado en EGFR, no mostraron ventaja de supervivencia en comparación con el placebo en la mayoría de los pacientes tratados (3,10). Sin embargo, un subgrupo de pacientes que incluía mujeres, no fumadoras, de origen asiático e histología de adenocarcinoma, probaron ser respondedoras al tratamiento con estos medicamentos (11-13). Los pacientes que respondieron a TKI presentaban mutaciones en los exones 18-21 del gen de EGFR. El 90% de estas mutaciones correspondían a pequeñas deleciones en el exón 19 o sustituciones de arginina por leucina en la posición 858 del exón 21 (14).
Dado el beneficio clínico de los TKIs, que se limita a un subgrupo específico de pacientes, existe necesidad en la identificación precisa de esta población. Varios estudios clínicos han sugerido que el número de copias del gen EGFR, la expresión de la proteína EGFR y las mutaciones activadoras en este gen, son prometedores biomarcadores predictivos de respuesta terapéutica al tratamiento con TKIs. Específicamente, se ha encontrado un aumento en el número de copias del gen EGFR, que generalmente da como resultado la sobreexpresión de la proteína de su mismo nombre en aproximadamente 30-40% de casos de NSCLC (15). Teóricamente, pacientes que tienen una mayor proporción de células cancerosas portadoras de mutaciones activadoras heterocigotas tienen una mayor tasa de respuesta a estos tratamientos. La abundancia de mutaciones de EGFR, ha sido documentada por la presencia de células portadoras de EGFR mutado y wild-type (16,17). Por ejemplo, Zhou y otros, sugirieron que los pacientes cuyos tumores tenían una gran abundancia de mutaciones de EGFR se beneficiaban más de TKIs que aquellos con una baja abundancia de las mismas. Además, se ha demostrado un número mayor de copias del gen EGFR en pacientes con mutaciones en comparación con mutaciones wild-type, lo que sugiere que la amplificación de EGFR podría ser la causa fundamental de la abundancia de mutantes.
Se han desarrollado múltiples técnicas para la identificación de amplificaciones de este marcador. Por otro lado, la hibridación fluorescente in situ (FISH) como predictor de respuesta a TKIs sigue siendo controvertida. Este estudio evaluó la asociación del estado de amplificación de EGFR detectado a través de FISH con varios resultados clínicos que incluyen la tasa de respuesta, la supervivencia global y la supervivencia libre de progresión en pacientes que reciben terapia con TKIs.
Materiales y Métodos
Este estudio incluyó a 72 pacientes con ADC pulmonar en estadio III o IV, portadores de mutaciones de EGFR tratados con Erlotinib como tratamiento de primera o segunda línea, en dos centros en Bogotá, Colombia (Fundación Santa Fe de Bogotá y Clínica del Country). El período de captación comenzó en enero de 2013 y finalizó en enero de 2016. Toda la información se recopiló en la plataforma-p de la Fundación para la Investigación Clínica y Aplicada del Cáncer (FICMAC, Bogotá, Colombia). Para asegurar la confidencialidad, un empleado independiente, eliminó los documentos de identificación personal del material histológico. Todos los datos clínicos se compararon y analizaron bajo codificación. Se revisó la historia clínica electrónica para obtener toda la información demográfica, clínica y patológica, bajo un protocolo aprobado por los comités de ética e investigación de las instituciones participantes. Los estudios de imagen fueron revisados independientemente por los autores para evaluar la respuesta al tratamiento de acuerdo con los Criterios de evaluación de respuesta en tumores sólidos (RECIST) versión 1.1 (18)
Preparación de las muestras
Las muestras de tejidos de pulmón fueron fijadas en formalina, embebidas en parafina (FFPE) durante el estudio patológico de rutina. Se eligieron bloques adecuados y se usaron cinco cortes de 10 \ mu m para la extracción de ADN. Se utilizó una lámina teñida con hematoxilina y eosina (H & E) de cada bloque para estimar el contenido de células tumorales. Todos los sujetos tenían suficiente tejido tumoral obtenido en el momento del diagnóstico primario mediante biopsia de cilindro (n = 52 / 72,0%) o toracoscopia / resección quirúrgica (n = 20 / 28,0%). Los sitios de muestreo fueron el tumor primario (n = 60 / 83,3%) o sitios metastásicos (n = 12 / 16,7%).Análisis con FISH del número de copias de EGFR
El análisis FISH se llevó a cabo utilizando la sonda LSI EGFR SpectrumOrange / CEP 7 SpectrumGreen (Vysis, Abbott Laboratories) de acuerdo con las indicaciones del fabricante. Las secciones se incubaron a 56°C durante la noche, se desparafinizaron mediante lavado en CitriSolv (Fisher Scientific, Pittsburgh, PA) y se deshidrataron en etanol al 100%. Después de la incubación en tampón de citrato sódico 2 × solución salina (2 × SSC, pH 7,0) a 75°C durante 15-25 minutos, las secciones se digirieron con proteinasa K (0,25 mg / ml en 2 × SSC, pH 7,0) a 37°C durante 15-25 minutos, se lavaron en 2 × SSC a temperatura ambiente durante 5 minutos y se deshidrataron en una serie de concentraciones crecientes de etanol (70%, 85% y 100%). El conjunto de sondas EGFR / CEP 7 se aplicó al área seleccionada basándose en la presencia de focos tumorales en cada portaobjetos, el área de hibridación se cubrió con un cubreobjetos de vidrio y se selló con esmalte de uñas. Los portaobjetos se incubaron a 80 ° C durante 8-10 minutos para la co-desnaturalización del ADN cromosómico y de la sonda. Luego se hibridaron a 37 ° C durante 20-24 horas. Se realizaron lavados posteriores a la hibridación en urea 1,5 M y SSC 0,1 × (pH 7,0 - 7,5) a 45 ° C durante 30 minutos, y en 2 x SSC durante 2 minutos a temperatura ambiente. Después de que las muestras se deshidrataran en etanol como se indicó anteriormente, se aplicó 4 ‘, 6’-diamidino-2-fenilindol (DAPI) en solución salina tamponada con fosfato y glicerol (Vysis) para la contratinción de la cromatina. Los análisis de FISH se realizaron de forma independiente por dos autores que estaban cegados a las características clínicas de los pacientes y a todas las demás variables moleculares. Para los análisis de FISH, se anotaron 60 núcleos para señales de ambas sondas de ADN usando un microscopio Olympus BX51TRF (Olympus, Tokio, Japón) equipado con un filtro de triple paso (DAPI / Verde / Naranja; Vysis) con un aumento final de 1000×. La polisomía y monosomía del cromosoma 7 se definieron como la presencia de ≥ tres señales y una señal, respectivamente, en más del 20% de las células tumorales. El estado del gen EGFR se clasificó en seis categorías según la frecuencia de células tumorales con números de copia específicos del gen EGFR y el centrómero del cromosoma 7: disomía (≤ 2 copias en ≥ 90% de las células), trisomía baja (≤ 2 copias en ≥ 40% de las células, 3 copias en 10 - 40% de las células y ≥ 4 copias en <10% de las células), trisomía alta (≤ 2 copias en ≥ 40% de las células, 3 copias en ≥ 40% de las células, y ≥ 4 copias en <10% de las células), polisomía baja (≥ 4 copias en 10 - 40% de las células), polisomía alta (≥ 4 copias en ≥ 40% de las células) y amplificación génica (a: presencia de grupos de genes EGFR estrechos (≥4 puntos) en ≥10% de células tumorales b: una relación del gen EGFR al cromosoma 7 de ≥ 2, c: ≥ 15 copias de EGFR por célula en ≥ 10% de células) (Figura suplementaria 1). En función del estado del gen EGFR, los pacientes se clasificaron en dos grupos: 1) EGFR negativa o baja (disomía, trisomía baja, trisomía alta y polisomía baja) y 2) EGFR positiva o alta (polisomía alta y amplificación génica). Para cada preparación de FISH, se usaron células conocidas positivas y negativas como controles.
Análisis de mutación EGFR
La prueba de tejido Cobas® EGFR (qPCR) se utilizó para la detección de mutaciones. Dicha prueba está diseñada para detectar sustituciones las G719A / C / S en el exón 18; 29 deleciones en el exón 19, 5 inserciones en el exón 20, entre ellas, S768I y T790M y la sustitución L858R en el exón 21. Además de las mutaciones detectadas en la prueba de tejido, se realizó análisis de sangre buscando la sustitución L861Q en el exón 21. Para las muestras FFPE, se usaron 50 ng de ADN genómico para cada reacción de PCR y para las muestras de plasma, Se usaron 25 μL del eluato de ADN para cada reacción de PCR. El software del sistema Cobas® 4800 SR2 v2.0 y el software de paquete de análisis EGFR v1.0 se usaron para el análisis de resultados.
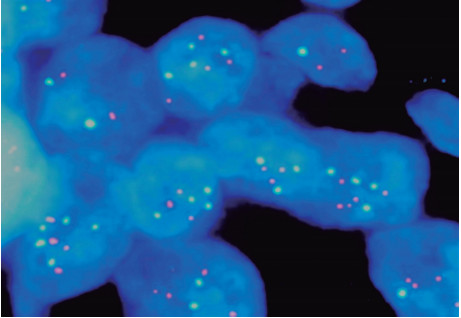
Figura Suplementaria 1. Cambio en el número de copias del gen del receptor del factor de crecimiento epidérmico (amplificación)
Análisis estadístico
Para fines descriptivos, las variables continuas se resumieron como medias aritméticas, medianas y desviaciones estándar. Las variables categóricas se informaron como proporciones con intervalos de confianza del 95% (IC del 95%). Las comparaciones se realizaron utilizando la prueba t de Student. χ2 o la prueba exacta de Fisher, se usaron para evaluar la significancia entre las variables categóricas. Las variables de tiempo evento, obtenidas del método de Kaplan-Meier, se determinaron mediante pruebas de log-rank. Para probar la asociación entre la expresión de ARNm (variables continuas) y las características clinicopatológicas (variables dicotómicas), se utilizaron las pruebas U de KruskalWallis y Mann-Whitney. La significancia estadística se determinó como p≤0,05 usando una prueba a dos colas. Todos los análisis estadísticos se realizaron con SPSS versión 19.0 (SPSS, Inc., Chicago, IL, EE. UU.).
Resultados
Características de los pacientes
Se seleccionaron un total de 72 pacientes adultos con adenocarcinoma de pulmón mutado EGFR, tratados con TKI en dos instituciones de Colombia. La Tabla 1 resume las características demográficas y clínicas de la población estudiada. La edad mediana en la muestra fue de 62 años (rango 20-87 años), 53 pacientes (73%) fueron mujeres, 46 (65%) tuvieron un puntaje de ECOG de 0-1, el sitio más común de afectación metastásica fue el hígado en 27 (37,5%) pacientes y el síntoma más frecuente fue tos crónica en 44 (61%) de los individuos muestreados. La minoría de la muestra (37,5%) tuvo un antecedente de tabaquismo positivo.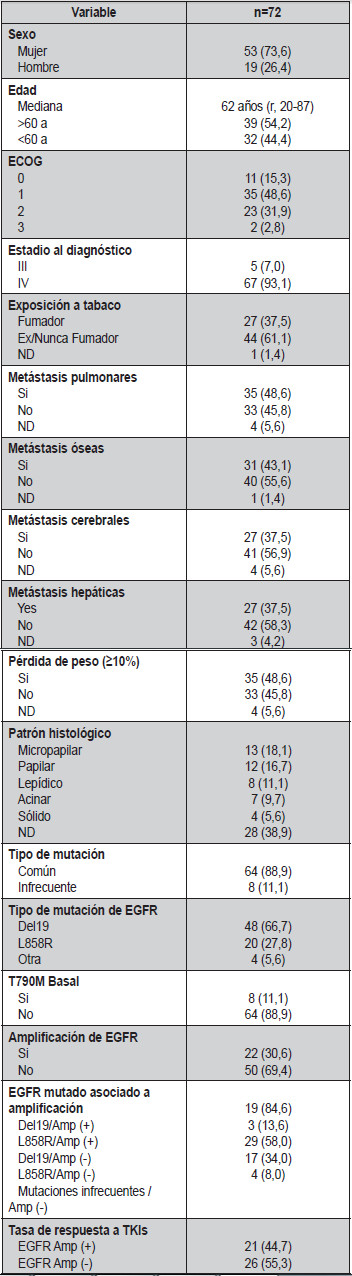
Tabla 1. Características de la cohorte
Características moleculares
De los 72 pacientes, el 89% albergaban la mutación Del19 (66,7%) o L858R (27,8%). También se identificaron un total de 22 (30,6%) pacientes con mutación concomitante de EGFR y amplificación con FISH. La amplificación se observó con mayor frecuencia en las muestras tumorales con Del19 en el 39,6% de los pacientes, en comparación con el 15% en el grupo de L858R (p = 0,05). También se asoció con una puntuación ECOG de 0-1 (p = 0,001) pero no se asoció con el tabaquismo (p = 0,85), con la edad superior a 60 años (p = 0,43) sexo (p = 0,2) o el patrón histológico (p = 0,71). Los perfiles moleculares se presentan en la Tabla 1.
Características del tratamiento, respuesta y análisis de supervivencia
La supervivencia libre de progresión (SLP) para la cohorte fue de 16,7 meses (IC95 11,1-22,3) (Figura 1).
42 pacientes (58,3%) recibieron quimioterapia como tratamiento de primera línea antes de la introducción de Erlotinib a la terapia estándar. Se observó una respuesta completa o parcial en 23 (54,8%) de los pacientes y se correlacionó con una mejor SLP, de 8,9 meses, en comparación con 5,13 meses en los que no respondieron (p = 0,003). Se observó respuesta al tratamiento con quimioterapia en el 36% de los pacientes con deleción del exón 19 en comparación con el 33,3% con mutación en el exón 21 (p = 0,057). En términos de SLP después de la quimioterapia, la mutación del EGFR también se asoció con mejores resultados, alcanzando 8,32 meses para el Exón 19, 5,83 meses para el exón 21 y 3,97 meses para otras mutaciones (p <0,001). La amplificación de EGFR se asoció con una mejor respuesta a la quimioterapia (p = 0,01), pero no con una mejor SLP después de esta (p = 0,18).
En términos de respuesta a Erlotinib, se observó una respuesta completa o parcial en 47 (65,3%) pacientes. La SLP en meses de respondedores a los TKIs fue de 27,5 en comparación con 7,43 en pacientes que no respondieron (p = 0,001). Cuando se estratificó por mutación, el 81,3% de los pacientes con mutaciones del exón 19 lograron respuesta terapéutica, mientras un 40% de los mutantes del exón 21 y el 0% de los pacientes con otras mutaciones (p = 0,001). Cuando se estratificó por mutación, la SLP arrojó una diferencia significativa entre el tipo de mutación (p <0,001) 27,5 meses para el Exón 19, 10,8 meses para el Exón 21 y 3,4 para otras mutaciones (Figura 2A). La amplificación de EGFR también reveló una diferencia en la mediana de SLP de 28,5 meses para las muestras amplificadas y 11 en las no amplificadas (p = 0,002) (Figura 2B). Al combinar la amplificación de EGFR y el tipo de mutación, el EGFR amplificado en el Exón 19 alcanzó una SLP similar en comparación con el EGFR amplificado en el Exón 21 (p = 0,166) (Figura 2C).
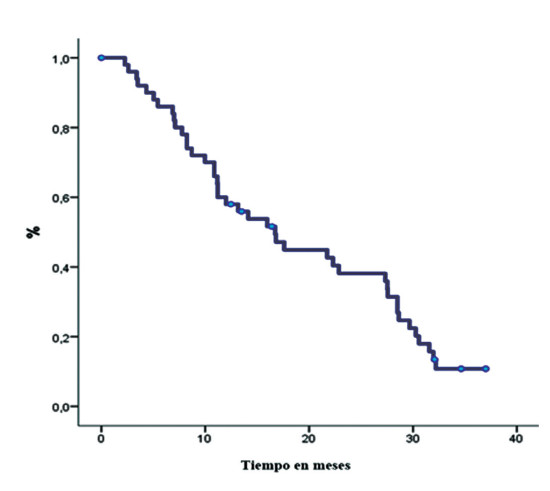
Figura 1. SLP general para la cohorte de pacientes estudiados
La mediana de supervivencia global (SG) fue de 32,9 meses (Figura 3).
Se observó una SG más larga en pacientes con mutaciones del Exón 19 (37,83 meses) en comparación con el Exón 21 (18,13 meses) y otras mutaciones (6,35 meses) (p = 0,001) (Figura 4A). De manera similar, la amplificación de EGFR también se asoció con una mejor OS (p = 0,009) (Figura 4B). La amplificación combinada de EGFR y la mutación del exón 19 ofrecieron una SG de 37,8 meses en comparación con 27,5 meses en el exón 21 amplificado de EGFR mutado (p <0,001) (figura 4C).
Discusión
Las mutaciones de los genes de la vía de señalización del receptor del factor de crecimiento epidérmico (EGFR) son mutaciones somáticas asociadas con pronóstico adverso en los cánceres de pulmón de células no pequeñas (NSLC). Comúnmente se pensaba que la activación de mutaciones somáticas en un alelo de un oncogen, es decir, una mutación heterocigótica (“un golpe”) era suficiente para la tumorigénesis (19,20). Sin embargo, otros mecanismos principales de la activación de oncogenes, “Copy Number Gains” (CNG) por su nombre y siglas en inglés, debido a la amplificación focal o polisomía cromosómica, se han relacionado con una mayor transcripción de alelos mutantes y por ende mayor actividad del gen de EGFR (21). La ganancia en el número de copias de este gen, está asociada con la activación de mutaciones solo en el locus oncogénico mutado, pero no en otros loci oncógenos (1). Este fenómeno ha sido descrito como desequilibrio específico del alelo mutante (MASI) causado por el aumento del número de copias o disomía uniparental (DUP) debido a la pérdida completa del alelo natural (20,21). Estas desregulaciones del gen EGFR, más la expresión de la proteína EGFR, se están estudiando como biomarcadores correlacionados con la respuesta al tratamiento con TKIs y la supervivencia en pacientes con NSCLC avanzado (15,22-25).
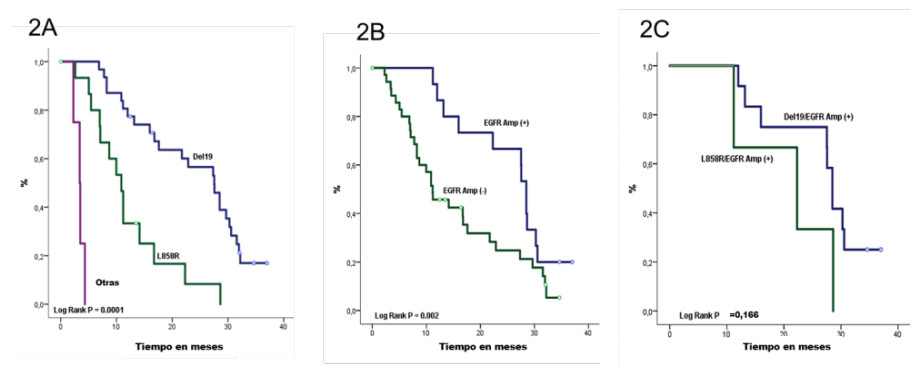
Figura
2. SLP según mutaciones y estado de amplificación de EGFR*
*2A) SLP según mutaciones de EGFR. La mutación EGFR en el Exón 19 (Deleción 19) tuvo una SLP de 27,5 meses (IC 95% 21,7-33,3), para el Exón 21 (L858R) la SSA fue de 10,8 meses (IC 95% 8,5-13,2) y para otras mutaciones 3,4 meses (IC 95% 2,1-4,6)
*2B) SLP según el estado de amplificación EGFR. Los pacientes con amplificación de EGFR (EGFR Amp (+)) mostraron una SLP de 28,5 meses (IC del 95%: 22,0-38,5), en contraste, pacientes sin amplificación de EGFR (Amp del EGFR (-) tuvieron una SSP de 11 meses (IC del 95%). 19,5).
*2C) SLP según la mutación de EGFR y el estado de amplificación. La SLP en pacientes con mutación y amplificación de EGFR (EGFR Mut / Amp (+)) con mutación del Exón 19 (Del19) fue de 28,5 meses (IC del 95% 26,9-30,0) y con la sustitución del Exón 21 (L858R) fue de 22,3 meses (95% CI 4,5-40,0). Los pacientes con mutación de EGFR y ausencia de amplificación (EGFR Mut / Amp (-)) mostraron una SLP de 21,7 meses (IC del 95%: 11,7-31,7) en los que tenían la mutación del exón 19 (Del19) y 8,7 meses (IC del 95%: 6,4- 15,8) en aquellos con sustitución Exón 21 (L858R).
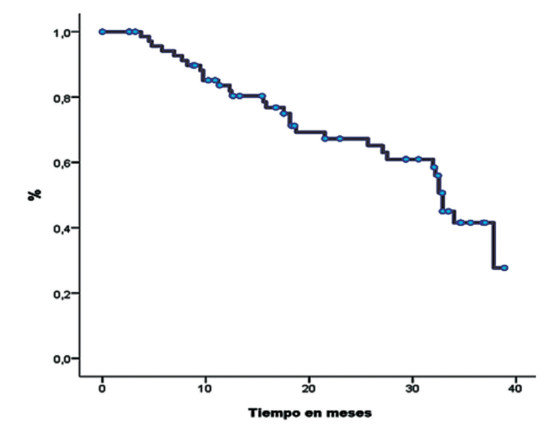
Figura 3. Mediana de SG para la cohorte de pacientes estudiados
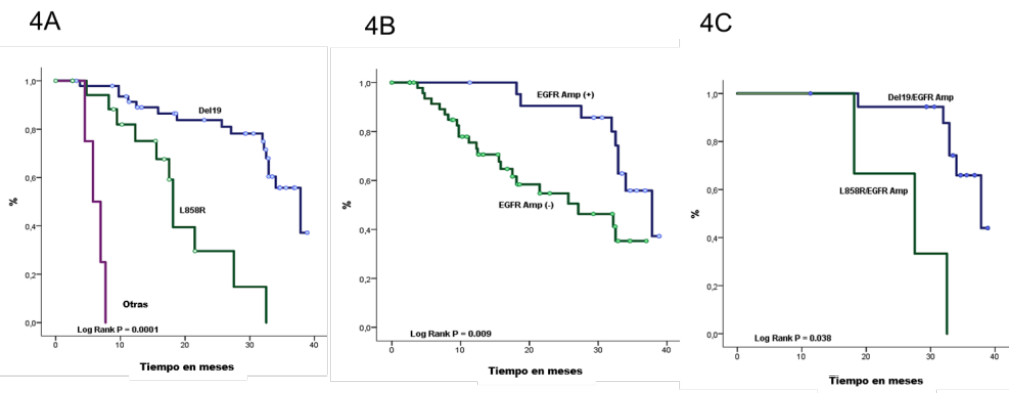
Figura 4. SG según la mutación y el estado de amplificación de EGFR*
*4A) SG
según la mutación EGFR. La mutación EGFR en el Exón 19 (Deleción 19)
tuvo una SG de 37,8
meses (IC 95% 31,2-44,4), para el Exón 21 (L858R) la SG fue de 18,1
meses (IC 95% 17,2-19,9)
y para otras mutaciones fue de 6,4 meses (95% CI 3,3-8,1).
*4B) SG según el estado de amplificación de EGFR. Los pacientes con amplificación de EGFR (EGFR Amp (+)) mostraron una SG de 37,8 meses (IC del 95%: 30,9-44,7); en cambio, los pacientes sin amplificación de EGFR (EGFR Amp (-)) tenían una SG de 27,1 meses (IC del 95%: 12,8 -41,3).
*4C) SG según la mutación EGFR y el estado de amplificación La SG en pacientes con mutación y amplificación de EGFR (EGFR Mut /Amp (+)) con mutación del Exón 19 (Del19) fue de 37,8 meses (IC 95% 31,0-44,6) y con la sustitución del Exón 21 (L858R) fue de 27,5 meses (95% CI 12,4-42,5). Los pacientes con mutación de EGFR y amplificación ausente (EGFR Mut / Amp (-)) presentaron una SG de 32,5 meses (IC del 95%: 18,6-34,7) en los que tenían la mutación del exón 19 (Del19) y 17,5 meses (IC del 95%: 14,6- 20,3) en aquellos con sustitución Exón 21 (L858R)
*4B) SG según el estado de amplificación de EGFR. Los pacientes con amplificación de EGFR (EGFR Amp (+)) mostraron una SG de 37,8 meses (IC del 95%: 30,9-44,7); en cambio, los pacientes sin amplificación de EGFR (EGFR Amp (-)) tenían una SG de 27,1 meses (IC del 95%: 12,8 -41,3).
*4C) SG según la mutación EGFR y el estado de amplificación La SG en pacientes con mutación y amplificación de EGFR (EGFR Mut /Amp (+)) con mutación del Exón 19 (Del19) fue de 37,8 meses (IC 95% 31,0-44,6) y con la sustitución del Exón 21 (L858R) fue de 27,5 meses (95% CI 12,4-42,5). Los pacientes con mutación de EGFR y amplificación ausente (EGFR Mut / Amp (-)) presentaron una SG de 32,5 meses (IC del 95%: 18,6-34,7) en los que tenían la mutación del exón 19 (Del19) y 17,5 meses (IC del 95%: 14,6- 20,3) en aquellos con sustitución Exón 21 (L858R)
Estos hallazgos son consistentes con estudios previos. Cappuzzo y otros, publicaron un estudio retrospectivo que muestra que los pacientes con NSCLC y tumores EGFR positivos para FISH (amplificación génica y polisomía alta), tuvieron tasas significativamente más altas de respuesta a Gefitinib (54% y 25%), SLP (9,1 y 6,3 meses) y tasas de supervivencia a 1 año (48,8% y 62,3%) más altas, que los pacientes con tumores FISH negativos, quienes sufrieron de enfermedad progresiva en más del 70%. El tiempo hasta la progresión fue de 3,5 meses o menos, y la mediana de supervivencia fue de 7 meses o menos (22). Por otra parte, en una revisión sistemática y de meta-análisis de Dahabreh et al., que incluyó 22 estudios con 1.821 pacientes (población caucásica y asiática), se describió que el aumento del número de copias del gen EGFR se asocia significativamente con una mejor SG (HR = 0,77; IC del 95%: 0,66-0,89; p = 0,001) y SLP (HR = 0,60; IC del 95%: 0,46-0,79; p <0,001) entre los pacientes tratados con TKIs (23). Finalmente, un metanálisis y una revisión sistemática recientes, realizados por Zhang et al., de 17 estudios con un total de 2.047 pacientes, demostraron que el aumento del número de copias del gen EGFR se asocia con una mayor tasa de respuesta y SLP en la población asiática y caucásica (p≤0,008), y una mejor SG en pacientes caucásicos (p≤0,018) (15). Los resultados de este estudio indican que el alto número de copias del gen EGFR identificado por FISH, puede ser un biomarcador eficaz para la sensibilidad a Erlotinib en pacientes hispanos con NSCLC avanzado.
Aunque las mutaciones de EGFR se consideran el primer paso en la tumorigénesis en el NSCLC, la presencia de células cancerosas que albergan tanto EGFR como mutaciones de EGFR, sugiere que estas dos alteraciones genéticas coexistentes, cooperan en el desarrollo del tumor (19,27). Además, la combinación de estos dos mecanismos activadores (mutación y MASI) puede conferir una mayor ventaja de crecimiento y supervivencia a la célula maligna que la presencia de un único mecanismo (20). En consecuencia, los tumores con ambos mecanismos activadores parecen ser más dependientes molecularmente de la vía EGFR y, debido a eso, podrían ser más sensibles a TKIs (19).
En este estudio se realizó un análisis de asociación entre el genotipo de la mutación EGFR, la amplificación de EGFR y los desenlaces del paciente, encontrando una diferencia estadística en la supervivencia (37,8 meses vs 27,5 meses) para la amplificación EGFR y la mutación Del19 o L858R, respectivamente (p <0,001). Sin embargo, la diferencia en SLP no alcanzó significación estadística (p = 0,116). Por lo tanto, en este estudio, la amplificación de EGFR para la mutación del exón 19 tuvo la mejor respuesta al tratamiento con EGFR-TKI. Además, los pacientes con mutación de sustitución L858R del exón 21 tuvieron una mayor mejoría en la supervivencia cuando la amplificación estaba presente, tanto para la SLP (22,3 meses frente a 8,7 meses) como para la SG (27,5 meses frente a 17,5 meses). Estos resultados son concordantes con el estudio retrospectivo realizado por Shan et al, que incluyó 86 pacientes con ADC pulmonar que albergaban mutaciones activadoras de EGFR y recibieron tratamiento con EGFR-TKI, encontraron en pacientes con deleciones en el exón 19, la mediana de SLP fue significativamente mayor en aquellos con amplificación de EGFR que aquellos sin (18,3 vs. 9,1 meses) (p = 0,021). Aunque no significativo, los pacientes con la mutación y la amplificación de L858R tuvieron una mediana de la SLP más larga, en comparación con aquellos sin amplificación (16,0 vs. 9,8 meses) (p = 0,178) (19).
Los hallazgos de este estudio son relevantes ya que siguieron a pacientes hispanos, mientras que la mayoría de los datos publicados se han limitado a la población caucásica y asiática. La etnicidad podría explicar la variabilidad genética y la capacidad predictiva del número de copias del gen EGFR en poblaciones que podrían albergar diferentes mutaciones somáticas de EGFR y amplificación de EGFR. Las limitaciones son su naturaleza retrospectiva y un tamaño de muestra pequeño. Además, no se incluyó la histología de células escamosas u otros factores coexistentes, como la expresión de la proteína EGFR y el estado de la mutación KRAS. También se sabe que estos factores afectan la respuesta de TKIs en NSCLC.
Conclusiones
Los estudios en diversas poblaciones permiten la expansión del conocimiento del paisaje genómico del NSCLC. Los resultados de este estudio respaldan la hipótesis, según la cual, el número de copias del gen EGFR detectado por FISH y las mutaciones sensibilizantes de EGFR, son biomarcadores asociados con una mejor supervivencia global, supervivencia libre de progresión y respuesta al tratamiento con EGFR-TKI en pacientes con NSCLC avanzado. En la búsqueda de herramientas predictivas y de pronóstico, se podría agregar el número de copia del gen EGFR como una nueva herramienta de diagnóstico, que permitirá el logro de un método de identificación preciso para pacientes con NSCLC avanzado que se beneficiarán con el tratamiento con TKIs.
Impacto Ambiental
Se dio manejo de residuos de análisis de histopatología, marcadores y demás reactantes de la manera estipulada por la normatividad vigente de manejo de residuos, establecido por la autoridad sanitaria competente. No se consideran otros impactos ambientales.
Conflicto de interés
Los autores declaran no tener ningún conflicto de interés.
Consideraciones Éticas
El estudio, al ser de carácter retrospectivo, en donde no hubo modificaciones en variables fisiológicas, psicológicas o sociales, se considera un estudio Sin Riesgo según el decreto 8430. Se dio protección y anonimización de datos personales en concordancia con la Ley de habeas data. Este estudio fue aprobado por el Comité de Ética e Investigación de las instituciones participantes.
Financiación de la investigación
El presente estudio ha sido respaldado por la Fundación para la Investigación Clínica y Aplicada del Cáncer - FICMAC (Bogotá, Colombia), Grant de investigación 017-2015.
Referencias
1. Molina JR, Yang P,
Cassivi SD, Schild SE, Adjei AA.
Non–Small Cell Lung Cancer: Epidemiology, Risk Factors, Treatment, and
Survivorship. Mayo Clin Proc Mayo
Clin. 2008 May; 83(5):584–94.
2. Lynch TJ, Bell DW, Sordella R, Gurubhagavatula S, Okimoto RA,
Brannigan BW, et al. Activating Mutations in
the Epidermal Growth Factor Receptor Underlying Responsiveness of
Non–Small-Cell Lung Cancer to Gefitinib. N Engl J Med. 2004 May 20;
350(21):2129–39.
3. Shepherd FA, Rodrigues Pereira J, Ciuleanu T, Tan EH,
Hirsh V, Thongprasert S, et al.
4. Erlotinib in Previously Treated Non–Small-Cell Lung
Cancer. N Engl J Med. 2005 Jul
5. 14; 353(2):123–32.
6. Tsao M-S, Sakurada A, Cutz J-C, Zhu C-Q, Kamel-Reid
S, Squire J, et al. Erlotinib in Lung
7. Cancer — Molecular and Clinical Predictors of Outcome.
N Engl J Med. 2005 Jul 14; 353(2):133–44.
8. Marchetti A, Martella C, Felicioni L, Barassi F, Salvatore
S, Chella A, et al. EGFR mutations in non-small-cell lung cancer:
analysis of a large series of cases and development of a rapid and
sensitive method for diagnostic
screening with potential implications on pharmacologic
treatment. J Clin Oncol Off J Am Soc Clin Oncol. 2005
Feb 1; 23(4):857–65.
9. Huang S-F, Liu H-P, Li L-H, Ku Y-C, Fu Y-N, Tsai H-Y, et
al. High frequency of epidermal growth factor receptor
mutations with complex patterns in non-small cell lung
cancers related to Gefitinib responsiveness in Taiwan.
Clin Cancer Res off J Am Assoc Cancer Res. 2004 Dec
15; 10(24):8195–203.
10. Arrieta O, Cardona AF, Martín C, Más-López L,
Corrales-Rodríguez L, Bramuglia G, et al. Updated Frequency
of EGFR and KRAS Mutations in NonSmall-Cell Lung
Cancer in Latin America: The Latin-American Consortium for the
Investigation of Lung Cancer (CLICaP). J
Thorac Oncol off Publ Int Assoc Study Lung Cancer.
2015 May; 10(5):838–43.
11. Arrieta O, Cardona AF, Federico Bramuglia G, Gallo A,
Campos-Parra AD, Serrano S, et al. Genotyping nonsmall cell lung cancer
(NSCLC) in Latin America. J Thorac Oncol off Publ Int Assoc Study Lung
Cancer. 2011
Nov; 6(11):1955–9.
12. Tokumo M, Toyooka S, Kiura K, Shigematsu H, Tomii K,
Aoe M, et al. The relationship between epidermal growth
factor receptor mutations and clinicopathologic features
in non-small cell lung cancers. Clin Cancer Res off J Am
Assoc Cancer Res. 2005 Feb 1; 11(3):1167–73.
13. Thatcher N, Chang A, Parikh P, Rodrigues Pereira J,
Ciuleanu T, von Pawel J, et al. Gefitinib plus best supportive care in
previously treated patients with refractory
advanced non-small-cell lung cancer: results from a randomised,
placebo-controlled, multicentre study (Iressa
Survival Evaluation in Lung Cancer). Lancet Lond Engl.
2005 Nov 29; 366(9496):1527–37.
14. Kris MG, Natale RB, Herbst RS, Lynch TJ, Prager D,
Belani CP, et al. Efficacy of Gefitinib, an inhibitor of the
epidermal growth factor receptor tyrosine kinase, in
symptomatic patients with non-small cell lung cancer: a
randomized trial. JAMA. 2003 Oct 22; 290(16):2149–58.
15. Fukuoka M, Yano S, Giaccone G, Tamura T, Nakagawa
K, Douillard J-Y, et al. Multi-institutional randomized phase II trial
of Gefitinib for previously treated patients with
advanced non-small-cell lung cancer (The IDEAL 1 Trial)
[corrected]. J Clin Oncol Off J Am Soc Clin Oncol. 2003
Jun 15; 21(12):2237–46.
16. Pérez-Soler R, Chachoua A, Hammond LA, Rowinsky
EK, Huberman M, Karp D, et al. Determinants of tumor
response and survival with Erlotinib in patients with nonsmall-cell
lung cancer. J Clin Oncol Off J Am Soc Clin
Oncol. 2004 Aug 15; 22(16):3238–47.
17. Santarpia M, Altavilla G, Salazar MF, Magri I, Pettineo
G, Benecchi S, et al. Tyrosine kinase inhibitors for nonsmall-cell lung
cancer: finding patients who will be responsive. Expert Rev Respir Med.
2011 Jun; 5(3):413–24.
18. Zhang X, Zhang Y, Tang H, He J. EGFR gene copy
number as a predictive/biomarker for patients with nonsmall-cell lung
cancer receiving tyrosine kinase inhibitor
treatment: a systematic review and meta-analysis. J Investig Med Off
Publ Am Fed Clin Res. 2017; 65(1):72–
81.
19. Taniguchi K, Okami J, Kodama K, Higashiyama M, Kato
K. Intratumor heterogeneity of epidermal growth factor
receptor mutations in lung cancer and its correlation
to the response to Gefitinib. Cancer Sci. 2008 May;
99(5):929–35.
20. Bai H, Wang Z, Wang Y, Zhuo M, Zhou Q, Duan J, et
al. Detection and Clinical Significance of Intratumoral
EGFR Mutational Heterogeneity in Chinese Patients
with Advanced Non-Small Cell Lung Cancer. Plos One.
2013 Feb 13; 8(2):e54170.
21. Eisenhauer EA, Therasse P, Bogaerts J, Schwartz LH,
Sargent D, Ford R, et al. New response evaluation criteria in solid
tumours: revised RECIST guideline (version
1.1). Eur J Cancer Oxf Engl 1990. 2009 Jan; 45(2):228–
47.
22. Shan L, Wang Z, Guo L, Sun H, Qiu T, Ling Y, et al.
Concurrence of EGFR amplification and sensitizing mutations indicate a
better survival benefit from EGFR-TKI
therapy in lung adenocarcinoma patients. Lung Cancer
Amst Neth. 2015 Sep; 89(3):337–42.
23. Gandhi J, Zhang J, Xie Y, Soh J, Shigematsu H, Zhang
W, et al. Alterations in genes of the EGFR signaling
pathway and their relationship to EGFR tyrosine kinase
inhibitor sensitivity in lung cancer cell lines. Plos One.
2009; 4(2):e4576.
24. Soh J, Okumura N, Lockwood WW, Yamamoto H, Shigematsu H, Zhang W,
et al. Oncogene mutations, copy
number gains and mutant allele specific imbalance
(MASI) frequently occur together in tumor cells. PloS
One. 2009 Oct 14; 4(10):e7464.
25. Cappuzzo F, Hirsch FR, Rossi E, Bartolini S, Ceresoli GL, Bemis
L, et al. Epidermal growth factor receptor gene and protein and
Gefitinib sensitivity in nonsmall-cell lung cancer. J Natl Cancer Inst.
2005 May 4;
97(9):643–55.
26. Dahabreh IJ, Linardou H, Kosmidis P, Bafaloukos D,
Murray S. EGFR gene copy number as a predictive
biomarker for patients receiving tyrosine kinase inhibitor treatment: a
systematic review and meta-analysis in
non-small-cell lung cancer. Ann Oncol off J Eur Soc Med
Oncol. 2011 Mar; 22(3):545–52.
27. Hirsch FR, Varella-García M, McCoy J, West H, Xavier
AC, Gumerlock P, et al. Increased epidermal growth
factor receptor gene copy number detected by fluorescence in situ
hybridization associates with increased sensitivity to Gefitinib in
patients with bronchioloalveolar
carcinoma subtypes: a Southwest Oncology Group Study. J Clin Oncol Off
J Am Soc Clin Oncol. 2005 Oct 1;
23(28):6838–45.
28. Hsu W-H, Yang JC-H, Mok TS, Loong HH. Overview
of current systemic management of EGFR-mutant NSCLC. Ann Oncol off J
Eur Soc Med Oncol. 2018 Jan 1;
29(suppl_1):i3–9.
29. Lee CK, Wu Y-L, Ding PN, Lord SJ, Inoue A, Zhou C,
et al. Impact of Specific Epidermal Growth Factor Receptor (EGFR)
Mutations and Clinical Characteristics on
Outcomes after Treatment with EGFR Tyrosine Kinase
Inhibitors versus Chemotherapy in EGFR-Mutant Lung
Cancer: A Meta-Analysis. J Clin Oncol Off J Am Soc Clin
Oncol. 2015 Jun 10; 33(17):1958–65.
30. Zhao Z-R, Wang J-F, Lin Y-B, Wang F, Fu S, Zhang S-L,
et al. Mutation abundance affects the efficacy of EGFR
tyrosine kinase inhibitor readministration in non-smallcell lung cancer
with acquired resistance. Med Oncol
Northwood Lond Engl. 2014 Jan; 31(1):810.
Recibido: 8 de febrero de 2019
Aceptado: 8 de mayo de 2019
Correspondencia:
Andrés Felipe Cardona
a_cardonaz@yahoo.com
Aceptado: 8 de mayo de 2019
Correspondencia:
Andrés Felipe Cardona
a_cardonaz@yahoo.com